Bevart sekvens - Conserved sequence

Sekvenser er aminosyrene for restene 120-180 av proteinene. Rester som er konservert på tvers av alle sekvenser er markert med grått. Under hvert sted (dvs. posisjon) for proteinsekvensjusteringen er en nøkkel som angir bevarte steder (*), steder med konservative erstatninger (:), steder med semi-konservative erstatninger (.) Og steder med ikke-konservative erstatninger () .
I evolusjonær biologi er konserverte sekvenser identiske eller lignende sekvenser i nukleinsyrer ( DNA og RNA ) eller proteiner på tvers av arter ( ortologe sekvenser ), eller i et genom ( paraloge sekvenser ), eller mellom donor og reseptor taxa ( xenologe sekvenser ). Bevaring indikerer at en sekvens har blitt opprettholdt av naturlig utvalg .
En svært konservert sekvens er en som har vært relativt uendret langt tilbake på fylogenetiske treet , og dermed langt tilbake i geologisk tid . Eksempler på høyt konserverte sekvenser inkluderer RNA-komponentene av ribosomer foreligger i alle områder av livet, homeobox sekvensene utbredt blant eukaryoter , og den tmRNA i bakterier . Studiet av sekvensbevaring overlapper feltene genomikk , proteomikk , evolusjonsbiologi , fylogenetikk , bioinformatikk og matematikk .
Historie
Oppdagelsen av DNAs rolle i arvelighet , og observasjoner av Frederick Sanger om variasjon mellom dyreinsuliner i 1949, fikk tidlige molekylærbiologer til å studere taksonomi fra et molekylært perspektiv. Studier på 1960-tallet brukte DNA-hybridisering og proteinkryssreaktivitetsteknikker for å måle likhet mellom kjente ortologe proteiner, for eksempel hemoglobin og cytokrom c . I 1965 introduserte Émile Zuckerkandl og Linus Pauling konseptet med den molekylære klokken , og foreslo at jevn erstatning av aminosyreutskiftning kunne brukes til å estimere tiden siden to organismer divergerte . Mens de innledende fylogeniene nært samsvarte med fossilrekorden , førte observasjoner om at noen gener syntes å utvikle seg med forskjellige hastigheter til utviklingen av teorier om molekylær evolusjon . Margaret Dayhoffs sammenligning av ferrodoksinsekvenser fra 1966 viste at naturlig seleksjon ville virke for å bevare og optimalisere proteinsekvenser som er viktige for livet.
Mekanismer
Over mange generasjoner kan nukleinsyresekvenser i genomet til en evolusjonær avstamning gradvis endres over tid på grunn av tilfeldige mutasjoner og slettinger . Sekvenser kan også rekombinere eller slettes på grunn av kromosomale omorganiseringer . Bevarte sekvenser er sekvenser som vedvarer i genomet til tross for slike krefter, og har lavere mutasjonshastigheter enn bakgrunnsmutasjonshastigheten.
Bevaring kan forekomme i kodende og ikke-kodende nukleinsyresekvenser. Svært konserverte DNA-sekvenser antas å ha funksjonell verdi, selv om rollen for mange høyt konserverte ikke-kodende DNA-sekvenser er dårlig forstått. I hvilken grad en sekvens bevares kan påvirkes av varierende utvalgstrykk , dens robusthet mot mutasjon, populasjonsstørrelse og genetisk drift . Mange funksjonelle sekvenser er også modulære og inneholder områder som kan være utsatt for uavhengig utvalgstrykk , for eksempel proteindomener .
Kodesekvens
I kodende sekvenser kan nukleinsyre- og aminosyresekvensen bevares i forskjellige omfang, ettersom degenerasjonen av den genetiske koden betyr at synonyme mutasjoner i en kodende sekvens ikke påvirker aminosyresekvensen til proteinproduktet.
Aminosyresekvenser kan bevares for å opprettholde strukturen eller funksjonen til et protein eller domene. Konserverte proteiner gjennomgår færre aminosyreerstatninger , eller er mer sannsynlig å erstatte aminosyrer med lignende biokjemiske egenskaper . I en sekvens kan aminosyrer som er viktige for bretting , strukturell stabilitet eller som danner et bindingssted, være mer konserverte.
Nukleinsyresekvensen til et proteinkodende gen kan også bevares av andre selektive trykk. Den kodonanvendelse skjevhet i enkelte organismer kan begrense typene av synonyme mutasjoner i en sekvens. Nukleinsyresekvenser som forårsaker sekundær struktur i mRNA i et kodende gen kan velges mot, ettersom noen strukturer kan påvirke translasjon negativt, eller konserveres der mRNA også fungerer som et funksjonelt, ikke-kodende RNA.
Ikke-koding
Ikke-kodende sekvenser som er viktige for genregulering , slik som bindings- eller gjenkjennelsessteder for ribosomer og transkripsjonsfaktorer , kan bevares i et genom. For eksempel kan promotoren til et konservert gen eller operon også bevares. Som med proteiner kan også nukleinsyrer som er viktige for strukturen og funksjonen til ikke-kodende RNA (ncRNA) bevares. Imidlertid er sekvensbevaring i ncRNA generelt dårlig sammenlignet med proteinkodende sekvenser, og basepar som bidrar til struktur eller funksjon blir ofte konservert i stedet.
Identifikasjon
Konserverte sekvenser identifiseres vanligvis ved bioinformatikkmetoder basert på sekvensjustering . Fremskritt innen DNA-sekvensering med høy gjennomstrømning og proteinmassespektrometri har vesentlig økt tilgjengeligheten av proteinsekvenser og hele genomer for sammenligning siden begynnelsen av 2000-tallet.
Homologisøk
Konserverte sekvenser kan identifiseres ved homologisøk , ved hjelp av verktøy som BLAST , HMMER , OrthologR og Infernal. Homologisøkingsverktøy kan ta en individuell nukleinsyre- eller proteinsekvens som input, eller bruke statistiske modeller generert fra flere sekvensjusteringer av kjente relaterte sekvenser. Statistiske modeller som profil-HMMer og RNA-kovariansemodeller som også inneholder strukturell informasjon, kan være nyttige når du søker etter fjernere relaterte sekvenser. Inngangssekvenser blir deretter justert mot en database med sekvenser fra beslektede individer eller andre arter. De resulterende justeringene blir deretter scoret basert på antall matchende aminosyrer eller baser, og antall hull eller slettinger generert av justeringen. Akseptable konservative substitusjoner kan identifiseres ved hjelp av substitusjonsmatriser som PAM og BLOSUM . Svært scorende justeringer antas å være fra homologe sekvenser. Bevaringen av en sekvens kan deretter utledes av påvisning av svært like homologer over et bredt fylogenetisk område.
Justering av flere sekvenser
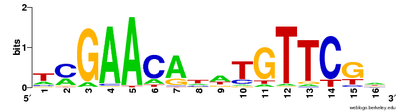
Flere sekvensjusteringer kan brukes til å visualisere konserverte sekvenser. Den CLUSTAL formatet omfatter en ren tekst nøkkel til å merke av konserverte kolonner av justeringen, betegner konservert sekvens (*), kan konservative mutasjoner (:), semi-konservative mutasjoner (.), Og ikke-konservative mutasjoner () Sekvens logoer også vise bevarte sekvens ved å representere proporsjonene av tegn på hvert punkt i justeringen etter høyde.
Genomjustering

Hele genomjusteringer (WGA) kan også brukes til å identifisere sterkt bevarte regioner på tvers av arter. For øyeblikket er nøyaktigheten og skalerbarheten til WGA -verktøy fortsatt begrenset på grunn av den beregningsmessige kompleksiteten ved å håndtere omorganiseringer, gjentatte regioner og den store størrelsen på mange eukaryote genomer. Imidlertid er WGAer med 30 eller mer nært beslektede bakterier (prokaryoter) nå stadig mer gjennomførbare.
Scoringssystemer
Andre tilnærminger bruker målinger av bevaring basert på statistiske tester som prøver å identifisere sekvenser som muterer annerledes enn en forventet (nøytral) mutasjonshastighet.
GERP (Genomic Evolutionary Rate Profiling) -rammeverket skårer bevaring av genetiske sekvenser på tvers av arter. Denne tilnærmingen anslår frekvensen av nøytral mutasjon i et sett med arter fra en flere sekvensjustering, og identifiserer deretter regioner i sekvensen som viser færre mutasjoner enn forventet. Disse regionene blir deretter tildelt poeng basert på forskjellen mellom den observerte mutasjonshastigheten og den forventede bakgrunnsmutasjonshastigheten. En høy GERP -score indikerer deretter en svært konservert sekvens.
LIST (Local Identity and Shared Taxa) er basert på antagelsen om at variasjoner observert hos arter som er nært knyttet til mennesker er mer signifikante når de vurderer bevaring sammenlignet med de hos arter som er langt i slekt. Dermed utnytter LIST den lokale justeringsidentiteten rundt hver posisjon for å identifisere relevante sekvenser i flersekvensjusteringen (MSA), og deretter estimerer den bevaring basert på taksonomiavstandene til disse sekvensene til mennesker. I motsetning til andre verktøy ignorerer LIST antallet/frekvensen av variasjoner i MSA.
Aminode kombinerer flere justeringer med fylogenetisk analyse for å analysere endringer i homologe proteiner og produsere et plott som indikerer de lokale evolusjonshastighetene. Denne tilnærmingen identifiserer de evolusjonært begrensede regionene i et protein, som er segmenter som er gjenstand for rensende utvalg og som vanligvis er kritiske for normal proteinfunksjon.
Andre tilnærminger som PhyloP og PhyloHMM inkorporerer statistiske fylogenetiske metoder for å sammenligne sannsynlighetsfordelinger av substitusjonshastigheter, noe som tillater påvisning av både bevaring og akselerert mutasjon. For det første genereres en bakgrunnssannsynlighetsfordeling av antall substitusjoner som forventes å skje for en kolonne i en justering med flere sekvenser, basert på et fylogenetisk tre . De estimerte evolusjonære forholdene mellom artene av interesse brukes til å beregne betydningen av eventuelle substitusjoner (dvs. en substitusjon mellom to nært beslektede arter kan være mindre sannsynlig å forekomme enn fjernt beslektede, og derfor mer signifikant). For å oppdage bevaring, beregnes en sannsynlighetsfordeling for en delmengde av flersekvensjusteringen, og sammenlignes med bakgrunnsfordelingen ved bruk av en statistisk test, for eksempel en sannsynlighetsforholdstest eller score-test . P-verdier generert ved sammenligning av de to fordelingene blir deretter brukt til å identifisere bevarte regioner. PhyloHMM bruker skjulte Markov -modeller for å generere sannsynlighetsfordelinger. PhyloP-programvarepakken sammenligner sannsynlighetsfordelinger ved hjelp av en sannsynlighetsforholdstest eller score-test , i tillegg til å bruke et GERP-lignende scoringssystem.
Ekstrem bevaring
Ultrakonserverte elementer
Ultrakonserverte elementer eller UCE er sekvenser som er svært like eller identiske på tvers av flere taksonomiske grupperinger . Disse ble først oppdaget hos virveldyr , og har senere blitt identifisert innenfor vidt forskjellige taxa. Mens opprinnelse og funksjon av UCES er dårlig forstått, har de blitt brukt til å undersøke dypt tidsavvik i amniotes , insekter , og mellom dyr og planter .
Universelt bevarte gener
De mest konserverte genene er de som finnes i alle organismer. Disse består hovedsakelig av ncRNA og proteiner som kreves for transkripsjon og oversettelse , som antas å ha blitt bevart fra den siste universelle felles forfaren til alt liv.
Gener eller genfamilier som har blitt funnet å være universelt konserverte inkluderer GTP-bindende forlengelsesfaktorer , metioninaminopeptidase 2 , serinhydroksymetyltransferase og ATP-transportører . Komponenter i transkripsjonsmaskineriet, slik som RNA -polymerase og helikaser , og i translasjonsmaskineriet, slik som ribosomale RNA , tRNA og ribosomale proteiner, er også universelt konservert.
applikasjoner
Filogenetikk og taksonomi
Sett med konserverte sekvenser brukes ofte for å generere fylogenetiske trær , da det kan antas at organismer med lignende sekvenser er nært beslektet. Valget av sekvenser kan variere avhengig av det taksonomiske omfanget av studien. For eksempel, de mest konserverte gener, slik som 16S RNA og andre ribosomale sekvenser som er nyttige for rekonstruksjon av dyp fylogenetiske forhold og identifisering av bakterielle phyla i metagenomikk studier. Sekvenser som er bevart i en klade, men som gjennomgår noen mutasjoner, for eksempel husholdningsgener , kan brukes til å studere artsforhold. Den interne transkriberte spacer -regionen (ITS), som er nødvendig for å holde avstand mellom bevarte rRNA -gener, men gjennomgår rask utvikling, brukes ofte til å klassifisere sopp og stammer av raskt utviklende bakterier.
Medisinsk forskning
Siden svært bevarte sekvenser ofte har viktige biologiske funksjoner, kan de være nyttige et utgangspunkt for å identifisere årsaken til genetiske sykdommer . Mange medfødte metabolske lidelser og lysosomale lagringssykdommer er et resultat av endringer i individuelle bevarte gener, noe som resulterer i manglende eller defekte enzymer som er den underliggende årsaken til symptomene på sykdommen. Genetiske sykdommer kan forutsies ved å identifisere sekvenser som er bevart mellom mennesker og laboratorieorganismer som mus eller fruktfluer , og studere effekten av knock-outs av disse genene. Genom-brede assosiasjonsstudier kan også brukes til å identifisere variasjon i bevarte sekvenser assosiert med sykdom eller helseutfall. Ved Alzehimers sykdom hadde det blitt funnet over to dusin nye potensielle sensitivitetslokaliteter
Funksjonell merknad
Identifisering av konserverte sekvenser kan brukes til å oppdage og forutsi funksjonelle sekvenser som gener. Konserverte sekvenser med en kjent funksjon, for eksempel proteindomener, kan også brukes til å forutsi funksjonen til en sekvens. Databaser over konserverte proteindomener som Pfam og Conserved Domain Database kan brukes til å kommentere funksjonelle domener i forutsagte proteinkodende gener.
Se også
- Evolusjonær utviklingsbiologi
- Segregerende nettsted
- Sekvensjustering
- Programvare for justering av sekvenser
- UCbase
- Ultrakonservert element
Referanser
