Ensembl genom databaseprosjekt - Ensembl genome database project
 | |
|---|---|
| Innhold | |
| Beskrivelse | Ensembl |
| Kontakt | |
| Forskningssenter | |
| Primær sitat | Yates, et al. (2020) |
| Adgang | |
| Nettsted | www |
Ensembl genomdatabaseprosjekt er et vitenskapelig prosjekt ved European Bioinformatics Institute , som ble lansert i 1999 som svar på den forestående gjennomføringen av Human Genome Project . Ensembl har som mål å gi en sentralisert ressurs for genetikere, molekylærbiologer og andre forskere som studerer genomene til vår egen art og andre virveldyr og modellorganismer . Ensembl er en av flere kjente genomnettlesere for gjenfinning av genomisk informasjon.
Lignende databaser og nettlesere finnes ved NCBI og University of California, Santa Cruz (UCSC) .
Bakgrunn
Det menneskelige genomet består av tre milliarder basepar , som koder for omtrent 20 000–25 000 gener . Imidlertid er genomet alene lite nyttig, med mindre stedene og forholdene til individuelle gener kan identifiseres. Ett alternativ er manuell kommentar , der et team av forskere prøver å finne gener ved hjelp av eksperimentelle data fra vitenskapelige tidsskrifter og offentlige databaser. Dette er imidlertid en langsom, møysommelig oppgave. Alternativet, kjent som automatisert annotering, er å bruke datamaskinens kraft til å gjøre den komplekse mønstertilpasningen av protein til DNA .
I Ensembl -prosjektet blir sekvensdata matet inn i genkommenteringssystemet (en samling programvare "pipelines" skrevet i Perl ) som lager et sett med forutsagte genlokasjoner og lagrer dem i en MySQL -database for senere analyse og visning. Ensembl gjør disse dataene fritt tilgjengelige for verdens forskningssamfunn. Alle dataene og koden produsert av Ensembl -prosjektet er tilgjengelig for nedlasting, og det er også en offentlig tilgjengelig databaseserver som tillater ekstern tilgang. I tillegg tilbyr Ensembl-nettstedet datagenererte visuelle visninger av mye av dataene.
Over tid prosjektet har utvidet til å omfatte flere arter (inkludert nøkkelmodellorganismer , slik som mus , bananflue og sebrafisk ), så vel som et bredere område av genomiske data, deriblant genetiske variasjoner og regulatoriske funksjoner. Siden april 2009 har et søsterprosjekt, Ensembl Genomes , utvidet omfanget av Ensembl til metazoa , planter , sopp , bakterier og protister , mens det opprinnelige prosjektet fortsetter å fokusere på virveldyr.
Viser genomiske data
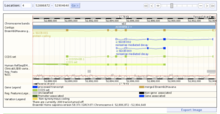
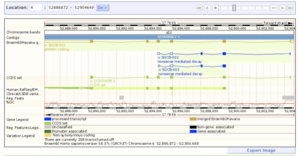
Sentralt i Ensembl -konseptet er muligheten til automatisk å generere grafiske visninger av justeringen av gener og andre genomiske data mot et referansegenom . Disse vises som dataspor, og individuelle spor kan slås av og på, slik at brukeren kan tilpasse skjermen slik at den passer til forskningsinteressene. Grensesnittet gjør det også mulig for brukeren å zoome inn på et område eller bevege seg langs genomet i begge retninger.
Andre skjermer viser data i varierende oppløsningsnivå, fra hele karyotyper til tekstbaserte representasjoner av DNA- og aminosyresekvenser , eller presenterer andre typer display som trær av lignende gener ( homologer ) på tvers av en rekke arter. Grafikken suppleres med tabelldisplayer, og i mange tilfeller kan data eksporteres direkte fra siden i en rekke standardfilformater som FASTA .
Eksternt produserte data kan også legges til i displayet ved å laste opp en passende fil i et av de støttede formatene, for eksempel BAM , BED eller PSL .
Grafikk genereres ved hjelp av en pakke med tilpassede Perl -moduler basert på GD , standard Perl -grafikkdisplaybibliotek.
Alternative tilgangsmetoder
I tillegg til nettstedet, tilbyr Ensembl et REST API og et Perl API (Application Programming Interface) som modellerer biologiske objekter som gener og proteiner, slik at enkle skript kan skrives for å hente data av interesse. Den samme APIen brukes internt av webgrensesnittet for å vise dataene. Den er delt inn i seksjoner som kjerne -API, compara API (for komparative genomiske data), variasjons -API (for tilgang til SNP, SNV, CNV ..) og funksjonell genomics API (for å få tilgang til regulatoriske data). Ensembl -nettstedet gir omfattende informasjon om hvordan du installerer og bruker API .
Denne programvaren kan brukes til å få tilgang til den offentlige MySQL -databasen, og unngå behovet for å laste ned enorme datasett. Brukerne kan til og med velge å hente data fra MySQL med direkte SQL -spørringer, men dette krever omfattende kunnskap om det nåværende databaseskjemaet.
Store datasett kan hentes ved hjelp av BioMart data-mining verktøyet. Det gir et webgrensesnitt for nedlasting av datasett ved hjelp av komplekse spørringer.
Sist er det en FTP -server som kan brukes til å laste ned hele MySQL -databaser samt noen utvalgte datasett i andre formater.
Nåværende art
De kommenterte genomene inkluderer de mest sekvenserte vertebrater og utvalgte modellorganismer. Alle er eukaryoter, det er ingen prokaryoter. Fra og med 2008 inkluderer dette:
-
Chordata
-
Mammalia
-
Euarkontoglires
- Primater : bushbaby , sjimpanse , menneske, makak , muselemur , orangutang , tarsier ;
- Scandentia : treskrue ;
- Glires (= Gnagere + Lagomorphs): guineapig , kenguru rotte , mus , rotte , bakkenekorn , pika , kanin ;
- Laurasiatheria : ku , delfin , alpakka , gris , katt , hund , hest , megabat , mikrobat , pinnsvin , spissmus ;
- Afrotheria: elefant , hyrax , tenrec
- Xenarthra : armadillo , dovendyr ;
- Marsupialia : opossum , wallaby ;
- Monotremes : platypus ;
-
Euarkontoglires
- Fugler : kylling , sebrafink ;
- Lepidosauria : anole firfirsle (pre);
- Lissamphibia : Xenopus tropicalis ;
- Teleostfisk : Takifugu rubripes ( fugu ), Tetraodon nigroviridis (grønn flekket pufferfish), Danio rerio ( sebrafisk ), Oryzias latipes ( medaka ), Gasterosteus aculeatus ( stickleback );
- Cyclostomata : Petromyzon marinus ( sea lamprey ) (pre);
- Tunikaer : Ciona intestinalis , Ciona savignyi ;
-
Mammalia
- Ikke-virveldyr
- Insekter : Drosophila melanogaster (fruktfugl), Anopheles gambiae (mygg), Aedes aegypti (mygg)
- Orm : Caenorhabditis elegans
- Gjær : Saccharomyces cerevisiae (bakergjær)
Se også
- Liste over sekvenserte eukaryote genomer
- Sekvensanalyse
- Sekvensprofileringsverktøy
- Sekvensmotiv
- UCSC Genome Browser
