Human Microbiome Project - Human Microbiome Project
| Human Microbiome Project (HMP) | |
|---|---|
 | |
| Eieren | US National Institutes of Health |
| Etablert | 2007 |
| Avviklet | 2016 |
| Nettsted | hmpdacc |
Den menneskelige microbiome Prosjekt ( HMP ) var en USA National Institutes of Health (NIH) forskning initiativ for å bedre forståelsen av de mikrobielle flora som er involvert i helse og sykdom. Den første fasen (HMP1) ble lansert i 2007 og fokuserte på å identifisere og karakterisere menneskelig mikrobiell flora. Den andre fasen, kjent som Integrative Human Microbiome Project (iHMP) ble lansert i 2014 med sikte på å generere ressurser for å karakterisere mikrobiomet og belyse mikrobenes rolle i helse- og sykdomstilstander. Programmet mottok 170 millioner dollar i finansiering fra NIH Common Fund fra 2007 til 2016.
Viktige komponenter i HMP var kulturuavhengige metoder for karakterisering av mikrobielt fellesskap , for eksempel metagenomics (som gir et bredt genetisk perspektiv på et enkelt mikrobielt samfunn), samt omfattende hele genom-sekvensering (som gir et "dypt" genetisk perspektiv på visse aspekter ved et gitt mikrobielt fellesskap, dvs. av individuelle bakteriearter). Sistnevnte tjente som referanse genomiske sekvenser - 3000 slike sekvenser for de enkelte bakterielle isolater er for tiden planlegges - for sammenligningsformål under etterfølgende metagenomic analyse. Prosjektet finansierte også dyp sekvensering av bakterielle 16S rRNA -sekvenser forsterket ved polymerasekjedereaksjon fra mennesker.
Introduksjon
Før HMP -lanseringen ble det ofte omtalt i populære medier og vitenskapelig litteratur at det er omtrent 10 ganger så mange mikrobielle celler og 100 ganger så mange mikrobielle gener i menneskekroppen som det er menneskelige celler; dette tallet var basert på estimater om at det menneskelige mikrobiomet inkluderer rundt 100 billioner bakterieceller og et voksent menneske har vanligvis rundt 10 billioner humane celler. I 2014 publiserte American Academy of Microbiology en FAQ som understreket at antall mikrobielle celler og antall menneskelige celler begge er estimater, og bemerket at nyere forskning hadde kommet frem til et nytt estimat av antall menneskelige celler på rundt 37 billioner celler , noe som betyr at forholdet mellom mikrobielle og menneskelige celler sannsynligvis er omtrent 3: 1. I 2016 publiserte en annen gruppe et nytt estimat på forholdet til å være omtrent 1: 1 (1,3: 1, med "en usikkerhet på 25% og en variasjon på 53% over befolkningen hos standard 70 kg hanner").
Til tross for det svimlende antallet mikrober i og på menneskekroppen, var lite kjent om deres roller i menneskers helse og sykdom. Mange av organismer som utgjør mikrobiomet har ikke blitt dyrket , identifisert eller på annen måte karakterisert. Organismer antatt å bli funnet i den humane mikrobiomer, men kan generelt kategoriseres som bakterier , medlemmer av domene Archaea , gjær og encellede eukaryote celler , så vel som forskjellige helmint parasitter og virus , sistnevnte innbefatter virus som infiserer de cellulære mikrobiomer organismene (f.eks. bakteriofager ). HMP satte seg for å oppdage og karakterisere det menneskelige mikrobiomet, med vekt på orale, hud-, vaginale, gastrointestinale og respiratoriske steder.
HMP vil ta for seg noen av de mest inspirerende, irriterende og grunnleggende vitenskapelige spørsmålene i dag. Viktigere er det også at det kan bryte ned de kunstige barrierer mellom medisinsk mikrobiologi og miljømikrobiologi. Det er håp at HMP ikke bare vil identifisere nye måter å bestemme helse og predisponering for sykdommer på, men også definere parametrene som trengs for å designe, implementere og overvåke strategier for forsettlig manipulering av den menneskelige mikrobiota, for å optimalisere ytelsen i sammenheng med individets fysiologi .
HMP har blitt beskrevet som "en logisk konseptuell og eksperimentell forlengelse av Human Genome Project ." I 2007 ble HMP oppført på NIH veikart for medisinsk forskning som en av de nye veiene til oppdagelse . Organisert karakterisering av det menneskelige mikrobiomet gjøres også internasjonalt i regi av International Human Microbiome Consortium. Den kanadiske Institutes of Health Research , gjennom CIHR Institute of Infection and Immunity, leder den kanadiske microbiome initiativ til å utvikle en koordinert og fokusert forskningsinnsats for å analysere og karakterisere mikrober som kolonisere menneskekroppen og deres potensielle endringer i løpet av kroniske sykdomstilstander.
Bidragende institusjoner
HMP involverte deltakelse fra mange forskningsinstitusjoner, inkludert Stanford University , Broad Institute , Virginia Commonwealth University , Washington University , Northeastern University , MIT , Baylor College of Medicine og mange andre . Bidragene inkluderte dataevaluering, konstruksjon av datasett for referansesekvenser, etiske og juridiske studier, teknologiutvikling og mer.
Fase én (2007-2014)
HMP1 inkluderte forskningsinnsats fra mange institusjoner. HMP1 satte seg følgende mål:
- Utvikle et referansesett med mikrobielle genom -sekvenser og utføre foreløpig karakterisering av det humane mikrobiomet
- Utforsk forholdet mellom sykdom og endringer i det menneskelige mikrobiomet
- Utvikle ny teknologi og verktøy for beregningsanalyse
- Etablere et ressursregister
- Studer de etiske, juridiske og sosiale implikasjonene av menneskelig mikrobiomforskning
Fase to (2014-2016)
I 2014 lanserte NIH den andre fasen av prosjektet, kjent som Integrative Human Microbiome Project (iHMP). Målet med iHMP var å produsere ressurser for å skape en fullstendig karakterisering av det menneskelige mikrobiomet, med fokus på å forstå tilstedeværelsen av mikrobiota i helse- og sykdomstilstander. Prosjektoppdraget, som nevnt av NIH, var som følger:
IHMP vil lage integrerte langsgående datasett med biologiske egenskaper fra både mikrobiomet og verten fra tre forskjellige kohortstudier av mikrobiomassosierte tilstander ved bruk av flere "omics" -teknologier.
Prosjektet omfattet tre delprosjekter som ble utført ved flere institusjoner. Studiemetoder inkluderte 16S rRNA -genprofilering, hel metagenome haglgeværsekvensering , helgenomsekvensering , metatranscriptomikk , metabolomikk / lipidomikk og immunoproteomikk . De viktigste funnene i iHMP ble publisert i 2019.
Graviditet og for tidlig fødsel
Vaginal Microbiome Consortium -teamet ved Virginia Commonwealth University ledet forskning på prosjektet Pregnancy & Preterm Birth med et mål om å forstå hvordan mikrobiomet endres i svangerskapsperioden og påvirker det nyfødte mikrobiomet. Prosjektet var også opptatt av mikrobiomets rolle i forekomsten av premature fødsler, som ifølge CDC står for nesten 10% av alle fødsler og utgjør den nest viktigste årsaken til nyfødt død. Prosjektet mottok 7,44 millioner dollar i NIH -finansiering.
Utbruddet av inflammatorisk tarmsykdom (IBD)
Inflammatory Bowel Disease Multi'omics Data (IBDMDB) -teamet var en gruppe med flere institusjoner som fokuserte på å forstå hvordan tarmmikrobiomet endres i lengderetningen hos voksne og barn som lider av IBD . IBD er en inflammatorisk autoimmun lidelse som manifesterer seg enten som Crohns sykdom eller ulcerøs kolitt og rammer omtrent en million amerikanere. Forskningsdeltakere inkluderte kohorter fra Massachusetts General Hospital , Emory University Hospital / Cincinnati Children's Hospital og Cedars-Sinai Medical Center .
Start av type 2 diabetes (T2D)
Forskere fra Stanford University og Jackson Laboratory of Genomic Medicine jobbet sammen for å utføre en langsgående analyse av de biologiske prosessene som forekommer i mikrobiomet til pasienter med risiko for type 2 diabetes . T2D påvirker nesten 20 millioner amerikanere med minst 79 millioner pasienter med diabetes før, og er delvis preget av markante endringer i mikrobiomet sammenlignet med friske individer. Prosjektet hadde som mål å identifisere molekyler og signalveier som spiller en rolle i etiologien til sykdommen.
Prestasjoner
Virkningen til dags dato av HMP kan delvis vurderes ved undersøkelse av forskning sponset av HMP. Over 650 fagfellevurderte publikasjoner ble oppført på HMP-nettstedet fra juni 2009 til slutten av 2017, og hadde blitt sitert over 70 000 ganger. På dette tidspunktet ble nettstedet arkivert og blir ikke lenger oppdatert, selv om datasett fortsatt er tilgjengelige.
Hovedkategorier av arbeid finansiert av HMP inkluderer:
- Utvikling av nye databasesystemer som muliggjør effektiv organisering, lagring, tilgang, søk og annotering av enorme datamengder. Disse inkluderer IMG, Integrated Microbial Genomes database og komparativt analysesystem; IMG/M, et beslektet system som integrerer metagenome datasett med isolerte mikrobielle genomer fra IMG -systemet; CharProtDB , en database med eksperimentelt karakteriserte proteinkommentarer; og Genomes OnLine Database (GOLD), for å overvåke statusen til genomiske og metagenomiske prosjekter over hele verden og tilhørende metadata.
- Utvikling av verktøy for komparativ analyse som letter gjenkjenning av vanlige mønstre, hovedtemaer og trender i komplekse datasett. Disse inkluderer RAPSearch2, et raskt og minneeffektivt proteinlikhetssøkverktøy for neste generasjons sekvenseringsdata; Boulder ALignment Editor (ALE), et nettbasert RNA-justeringsverktøy; WebMGA, en tilpassbar webserver for rask metagenomisk sekvensanalyse; og DNACLUST, et verktøy for nøyaktig og effektiv gruppering av fylogenetiske markørgener
- Utvikling av nye metoder og systemer for montering av massive sekvensdatasett. Ingen enkeltmonteringsalgoritme løser alle de kjente problemene med å sette sammen korte sekvenser, så neste generasjons monteringsprogrammer som AMOS er modulære og tilbyr et bredt spekter av verktøy for montering. Nye algoritmer er utviklet for å forbedre kvaliteten og bruken av utkast til genom -sekvenser.
- Montering av en katalog med sekvenserte referansegenomer av rene bakteriestammer fra flere kroppssteder, mot hvilke metagenomiske resultater kan sammenlignes. Det opprinnelige målet om 600 genomer er langt overgått; det nåværende målet er at 3000 genomer skal være i denne referansekatalogen, sekvensert til minst et utkaststrinn av høy kvalitet. Fra mars 2012 har 742 genomer blitt katalogisert.
- Etablering av Data Analysis and Coordination Center (DACC), som fungerer som det sentrale depotet for alle HMP -data.
- Ulike studier som utforsker juridiske og etiske problemstillinger knyttet til helgenomsekvensforskning.
Utviklingen finansiert av HMP inkluderte:
- Nye prediktive metoder for å identifisere aktive transkripsjonsfaktorbindingssteder.
- Identifikasjon, på grunnlag av bioinformatisk bevis, for en vidt distribuert, ribosomalt produsert elektronbærerforløper
- Time-lapse "bevegelige bilder" av det menneskelige mikrobiomet.
- Identifikasjon av unike tilpasninger adoptert av segmenterte filamentøse bakterier (SFB) i deres rolle som tarmkommensaler. SFB er medisinsk viktige fordi de stimulerer T -hjelper 17 -celler , antatt å spille en nøkkelrolle ved autoimmun sykdom .
- Identifikasjon av faktorer som skiller mikrobiotaen til frisk og syk tarm.
- Identifikasjon av en hittil ukjent dominerende rolle som Verrucomicrobia i jordbakteriesamfunn.
- Identifikasjon av faktorer som bestemmer virulenspotensialet til Gardnerella vaginalis -stammer ved vaginose .
- Identifikasjon av en kobling mellom oral mikrobiota og åreforkalkning.
- Demonstrasjon om at patogene arter av Neisseria er involvert i hjernehinnebetennelse , sepsis og seksuelt overførbare sykdommer utveksler virulensfaktorer med kommensale arter.
Milepæler
Referansedatabase etablert
Juni 2012 ble en stor milepæl for HMP kunngjort av NIH -direktøren Francis Collins . Kunngjøringen ble ledsaget av en serie koordinerte artikler publisert i Nature og flere tidsskrifter, inkludert Public Library of Science (PLoS) samme dag. Ved å kartlegge den normale mikrobielle sammensetningen av friske mennesker ved hjelp av genom-sekvenseringsteknikker, har forskerne ved HMP opprettet en referansedatabase og grensene for normal mikrobiell variasjon hos mennesker.
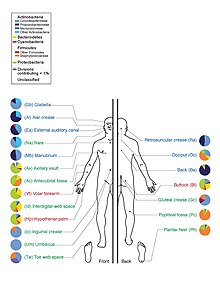
Fra 242 friske amerikanske frivillige ble mer enn 5000 prøver samlet fra vev fra 15 (menn) til 18 (kvinner) kroppssteder som munn, nese, hud, nedre tarm (avføring) og skjede. Alt DNA, humant og mikrobielt, ble analysert med DNA -sekvenseringsmaskiner. De mikrobielle genomdataene ble ekstrahert ved å identifisere det bakteriespesifikke ribosomale RNA, 16S rRNA . Forskerne beregnet at mer enn 10 000 mikrobielle arter okkuperer det menneskelige økosystemet, og de har identifisert 81 - 99% av slektene . I tillegg til å etablere den menneskelige mikrobiomreferansedatabasen, oppdaget HMP -prosjektet også flere "overraskelser", som inkluderer:
- Mikrober bidrar med flere gener som er ansvarlige for menneskelig overlevelse enn menneskers egne gener. Det er anslått at bakterielle proteinkodende gener er 360 ganger mer rikt enn menneskelige gener.
- Mikrobielle metabolske aktiviteter; for eksempel fordøyelse av fett; blir ikke alltid levert av de samme bakterieartene. Tilstedeværelsen av aktivitetene ser ut til å ha større betydning.
- Komponenter i det humane mikrobiomet endres over tid, påvirket av pasientens sykdomstilstand og medisinering. Imidlertid går mikrobiomet til slutt tilbake til en likevektstilstand, selv om sammensetningen av bakterietyper har endret seg.
Klinisk anvendelse
Blant de første kliniske applikasjonene som benyttet HMP -dataene, som rapportert i flere PLoS -artikler, fant forskerne et skifte til mindre artsmangfold i vaginalt mikrobiom hos gravide som forberedelse til fødsel, og høy viral DNA -belastning i nesemikrobiomet hos barn med uforklarlig feber. Andre studier som bruker HMP -data og teknikker inkluderer mikrobiomets rolle i ulike sykdommer i fordøyelseskanalen, huden, reproduktive organer og barndomsforstyrrelser.
Farmasøytisk anvendelse
Farmasøytiske mikrobiologer har vurdert implikasjonene av HMP-dataene i forhold til tilstedeværelse / fravær av 'anstøtelige' mikroorganismer i ikke-sterile farmasøytiske produkter og i forhold til overvåking av mikroorganismer i de kontrollerte miljøene der produktene produseres. Sistnevnte har også implikasjoner for medievalg og desinfiserende effektstudier.
Se også
Referanser
Eksterne linker
- Human Microbiome Project
- Dataanalyse og koordinasjonssenter
- CIHR Canadian Microbiome Initiative
- International Human Microbiome Consortium
- 2006, Lay -sammendrag av kolonmikrobiomstudie ( den faktiske studien: Gill et al., 2006 )
- Olivia Judson Microbes 'R' Us New York Times 22. juli 2009
- Gina Kolata God helse? Takk dine 100 billioner bakterier New York Times 13. juni 2012