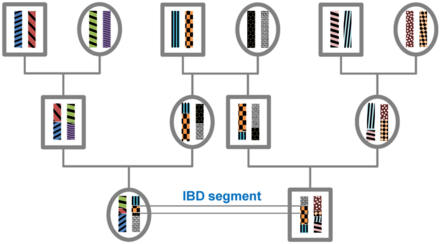
Identitet etter herkomst - Identity by descent
Et DNA -segment er identisk etter tilstand (IBS) i to eller flere individer hvis de har identiske nukleotidsekvenser i dette segmentet. Et IBS -segment er identisk ved nedstigning ( IBD ) i to eller flere individer hvis de har arvet det fra en felles stamfar uten rekombinasjon , det vil si at segmentet har samme forfedre opprinnelse hos disse individene. DNA -segmenter som er IBD er IBS per definisjon, men segmenter som ikke er IBD kan fortsatt være IBS på grunn av de samme mutasjonene i forskjellige individer eller rekombinasjoner som ikke endrer segmentet.
Teori
Alle individer i en begrenset befolkning er i slekt hvis de spores tilbake lenge nok og vil derfor dele deler av genomene IBD. Under meiose brytes segmenter av IBD opp ved rekombinasjon. Derfor er den forventede lengden på et IBD -segment avhengig av antall generasjoner siden den siste vanlige stamfaren på segmentet. Lengden på IBD -segmenter som er et resultat av en felles stamfar n generasjoner tidligere (derfor involvert 2 n meiose) er eksponentielt fordelt med gjennomsnittlig 1/(2 n ) Morgans (M). Det forventede antallet IBD -segmenter avtar med antall generasjoner siden den felles forfaren på dette stedet. For et spesifikt DNA -segment reduseres sannsynligheten for å være IBD som 2 - 2 n siden sannsynligheten for å overføre dette segmentet i hver meiose er 1/2.
applikasjoner
Identifiserte IBD -segmenter kan brukes til en rekke formål. Som nevnt ovenfor avhenger mengden (lengde og antall) av IBD -deling av de familiære forholdene mellom de testede individene. Derfor er en anvendelse av IBD -segmentdeteksjon å kvantifisere beslektethet. Måling av beslektethet kan brukes i rettsmedisinsk genetikk , men kan også øke informasjonen i kartlegging av genetisk kobling og bidra til å redusere skjevhet ved udokumenterte forhold i standard foreningsstudier . En annen anvendelse av IBD er genotype -imputasjon og haplotype fase -slutning. Lange delte segmenter av IBD, som er delt opp i korte regioner, kan indikere fasefeil.
IBD -kartlegging
IBD -kartlegging ligner koblingsanalyse, men kan utføres uten kjent stamtavle på en kohorte av ikke -relaterte individer. IBD -kartlegging kan sees på som en ny form for assosiasjonsanalyse som øker kraften til å kartlegge gener eller genomiske regioner som inneholder flere varianter av sjeldne sykdommer.
Ved å bruke simulerte data, viste Browning og Thompson at IBD -kartlegging har høyere effekt enn assosiasjonstesting når flere sjeldne varianter i et gen bidrar til sykdomsfølsomhet. Via IBD-kartlegging ble signifikante regioner i genomet i isolerte populasjoner så vel som avlede populasjoner funnet mens standard assosiasjonstester mislyktes. Houwen et al. brukte IBD -deling for å identifisere den kromosomale plasseringen av et gen som er ansvarlig for godartet tilbakevendende intrahepatisk kolestase i en isolert fiskebestand. Kenny et al. brukte også en isolert befolkning til å kartlegge et signal som ble funnet av en genomomfattende assosiasjonsstudie (GWAS) av plasmaplantesterolnivåer (PPS), et surrogatmål for kolesterolabsorpsjon fra tarmen. Francks et al. klarte å identifisere et potensielt mottakelighetslokus for schizofreni og bipolar lidelse med genotypedata fra case-control-prøver. Lin et al. fant et signifikant koblingssignal på hele genomet i et datasett for pasienter med multippel sklerose . Letouzé et al. brukte IBD -kartlegging for å lete etter grunnleggende mutasjoner i kreftprøver .
IBD i populasjonsgenetikk
Deteksjon av naturlig seleksjon i det menneskelige genomet er også mulig via påviste IBD -segmenter. Utvalg vil vanligvis ha en tendens til å øke antallet IBD -segmenter blant individer i en befolkning. Ved å søke etter regioner med overdreven IBD -deling, kan regioner i det menneskelige genomet som har vært under sterkt, svært nylig utvalg identifiseres.
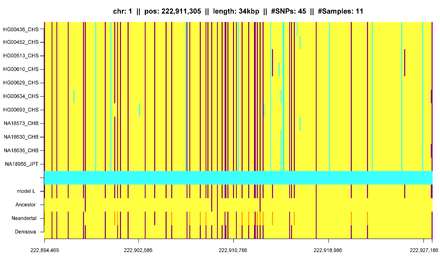
I tillegg til det kan IBD -segmenter være nyttige for å måle og identifisere andre påvirkninger på befolkningsstrukturen. Gusev et al. viste at IBD -segmenter kan brukes med tilleggsmodellering for å estimere demografisk historie inkludert flaskehalser og blanding . Ved bruk av lignende modeller Palamara et al. og Carmi et al. rekonstruerte den demografiske historien til Ashkenazi -jødiske og kenyanske masai -individer . Botigué et al. undersøkte forskjeller i afrikansk aner blant europeiske befolkninger. Ralph og Coop brukte IBD -deteksjon for å kvantifisere felles opphav til forskjellige europeiske populasjoner og Gravel et al. prøvde på samme måte å trekke konklusjoner om den genetiske historien til populasjoner i Amerika. Ringbauer et al. benyttet den geografiske strukturen til IBD -segmenter for å estimere spredning i Øst -Europa i løpet av de siste århundrene. Ved å bruke 1000 genomdataene fant Hochreiter forskjeller i IBD -deling mellom afrikansk, asiatisk og europeisk befolkning samt IBD -segmenter som deles med gamle genomer som neandertaleren eller Denisova .
Metoder og programvare
Programmer for påvisning av IBD -segmenter hos ikke -relaterte individer:
- RAPID : Ultra-fast Identity by Descent Detection in Biobank-Scale Cohorts using Positional Burrows-Wheeler Transform
- Foreldre : identifiserer IBD -segmenter mellom par av individer i ikke -vektede genotypedata
- BEAGLE/fastIBD : finner segmenter av IBD mellom par av individer i genomdekkende SNP- data
- BEAGLE/RefinedIBD : finner IBD -segmenter i par av individer som bruker en hashing -metode og evaluerer deres betydning via et sannsynlighetsforhold
- IBDseq : oppdager parvise IBD -segmenter i sekvenseringsdata
- GERMLINE : oppdager i lineære IBD-segmenter i par med individer
- DASH : bygger på parvise IBD -segmenter for å utlede klynger av individer som sannsynligvis vil dele en enkelt haplotype
- PLINK : er et verktøysett for hele genomforening og populasjonsbaserte koblingsanalyser, inkludert en metode for parvis IBD-segmentdeteksjon
- Relatere : estimerer sannsynligheten for IBD mellom par av individer på et bestemt sted ved hjelp av SNP -er
- MCMC_IBDfinder : er basert på Markov -kjeden Monte Carlo (MCMC) for å finne IBD -segmenter hos flere individer
- IBD-Groupon : oppdager gruppemessige IBD-segmenter basert på parvise IBD-forhold
- HapFABIA : identifiserer svært korte IBD -segmenter preget av sjeldne varianter i store sekvenseringsdata samtidig hos flere individer
Se også
- Foreningskartlegging
- Genetisk forening
- Genetisk kobling
- Genomeomfattende foreningsstudie
- Identitet etter type
- Kobling ulikhet
- Befolkningsgenetikk