Nukleosom - Nucleosome

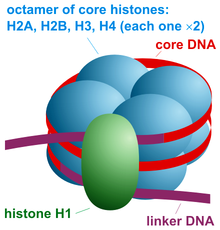
Et nukleosom er den grunnleggende strukturelle enheten for DNA -emballasje i eukaryoter . Strukturen til et nukleosom består av et segment av DNA som er viklet rundt åtte histonproteiner og ligner tråd viklet rundt en spole. Nukleosomet er den grunnleggende underenheten til kromatin . Hvert nukleosom består av litt mindre enn to omdreininger av DNA viklet rundt et sett med åtte proteiner kalt histoner, som er kjent som en histonoktamer . Hver histonoktamer består av to kopier hver av histonproteinene H2A , H2B , H3 og H4 .
DNA må komprimeres til nukleosomer for å passe inn i cellekjernen . I tillegg til nukleosominnpakning, komprimeres eukaryotisk kromatin ytterligere ved å brettes til en rekke mer komplekse strukturer og til slutt danne et kromosom . Hver menneskelig celle inneholder omtrent 30 millioner nukleosomer.
Nukleosomer antas å bære epigenetisk arvet informasjon i form av kovalente modifikasjoner av kjernehistonene . Nukleosomposisjoner i genomet er ikke tilfeldige, og det er viktig å vite hvor hvert nukleosom er plassert fordi dette bestemmer tilgjengeligheten til DNA for regulatoriske proteiner .
Nukleosomer ble først observert som partikler i elektronmikroskopet av Don og Ada Olins i 1974, og deres eksistens og struktur (som histonoktamerer omgitt av omtrent 200 basepar DNA) ble foreslått av Roger Kornberg . Nukleosomets rolle som en generell genrepressor ble demonstrert av Lorch et al. in vitro, og av Han og Grunstein in vivo henholdsvis 1987 og 1988.
Nucleosome kjernepartikler som består av omtrent 146 basepar (bp) av DNA pakket i 1,67 venstrehendte superheliksform vindinger rundt en histon oktamer, bestående av 2 eksemplarer hver av kjernen histoner H2A , H2B , H3 og H4 . Kjernepartikler er forbundet med strekninger av linker -DNA , som kan være opptil 80 bp lange. Teknisk sett er et nukleosom definert som kjernepartikkelen pluss en av disse linkerregionene; Imidlertid er ordet ofte synonymt med kjernepartikkelen. Genome-wide nukleosomposisjoneringskart er nå tilgjengelig for mange modellorganismer, inkludert muselever og hjerne.
Linkerhistoner som H1 og dets isoformer er involvert i kromatinkomprimering og sitter ved basen av nukleosomet nær DNA -inngangen og utgangsbinding til linkerområdet til DNA. Ikke-kondenserte nukleosomer uten linkerhiston ligner "perler på en DNA-streng" under et elektronmikroskop .
I motsetning til de fleste eukaryote celler bruker modne sædceller i stor grad protaminer for å pakke sitt genomiske DNA, mest sannsynlig oppnå et enda høyere emballasjeforhold. Histonekvivalenter og en forenklet kromatinstruktur er også funnet i Archaea , noe som antyder at eukaryoter ikke er de eneste organismer som bruker nukleosomer.
Struktur
Struktur av kjernepartikkelen
Oversikt
Banebrytende strukturstudier på 1980-tallet av Aaron Klugs gruppe ga det første beviset på at en oktamer av histonproteiner vikler DNA rundt seg selv i omtrent 1,7 svinger av en venstrehendt superhelix. I 1997 ble den første nær atomoppløselige krystallstrukturen til nukleosomet løst av Richmond -gruppen, som viste de viktigste detaljene i partikkelen. Det menneskelige alfa -satellitt -palindromiske DNA som er kritisk for å oppnå nukleosomkrystallstrukturen fra 1997 ble utviklet av Bunick -gruppen ved Oak Ridge National Laboratory i Tennessee. Strukturene til over 20 forskjellige nukleosomkjernepartikler er løst til dags dato, inkludert de som inneholder histonvarianter og histoner fra forskjellige arter. Strukturen til nukleosomkjernepartikkelen er bemerkelsesverdig bevart, og til og med en endring på over 100 rester mellom frosk- og gjærhistoner resulterer i elektrontetthetskart med en total gjennomsnittlig kvadratisk avvik på rot på bare 1,6 Å.
Nukleosomkjernepartikkelen (NCP)
Nukleosomkjernepartikkelen (vist på figuren) består av omtrent 146 basepar DNA innpakket i 1,67 venstrehendte superheliske svinger rundt histonoktameren , bestående av 2 kopier hver av kjernehistonene H2A , H2B , H3 og H4 . Tilstøtende nukleosomer er forbundet med en strekning av fritt DNA kalt linker -DNA (som varierer fra 10 til 80 bp i lengde avhengig av art og vevstype). Hele strukturen genererer en sylinder med en diameter på 11 nm og en høyde på 5,5 nm.

Nukleosomkjernepartikler observeres når kromatin i interfase behandles for å få kromatinet til å utfolde seg delvis. Det resulterende bildet, via et elektronmikroskop, er "perler på en streng". Strengen er DNA, mens hver perle i nukleosomet er en kjernepartikkel. Nukleosomkjernepartikkelen består av DNA og histonproteiner.
Delvis DNA -fordøyelse av kromatin avslører dens nukleosomstruktur. Fordi DNA -deler av nukleosomkjernepartikler er mindre tilgjengelige for DNAse enn koblingsseksjoner, blir DNA fordøyd til fragmenter av lengder som er lik avstanden mellom nukleosomer (180, 360, 540 basepar osv.). Derfor er et veldig karakteristisk mønster som ligner på en stige synlig under gelelektroforese av det DNA. Slik fordøyelse kan også forekomme under naturlige forhold under apoptose (" cellemord " eller programmert celledød), fordi autodestruksjon av DNA vanligvis er dens rolle.
Proteininteraksjoner i nukleosomet
Kjernehistonproteinene inneholder et karakteristisk strukturmotiv kalt "histonfolden", som består av tre alfa-helixer (α1-3) atskilt med to sløyfer (L1-2). I løsning danner histonene H2A-H2B heterodimerer og H3-H4 heterotetramere. Histoner dimererer om sine lange α2-helixer i en antiparallell orientering, og i tilfelle av H3 og H4 danner to slike dimerer en 4-helix-bunt stabilisert av omfattende H3-H3 '-interaksjon. H2A/H2B -dimeren binder seg til H3/H4 -tetrameren på grunn av interaksjoner mellom H4 og H2B, som inkluderer dannelse av en hydrofob klynge. Histonoktameren dannes av en sentral H3/H4 -tetramer klemt mellom to H2A/H2B -dimerer. På grunn av den svært grunnleggende ladningen til alle de fire kjernehistonene, er histonoktameret stabilt bare i nærvær av DNA eller svært høye saltkonsentrasjoner.
Histon - DNA -interaksjoner
Nukleosomet inneholder over 120 direkte protein-DNA-interaksjoner og flere hundre vannmedierte. Direkte protein - DNA -interaksjoner spres ikke jevnt om oktameroverflaten, men ligger på diskrete steder. Disse skyldes dannelsen av to typer DNA -bindingssteder i oktameren; α1α1 -stedet, som bruker α1 -helixen fra to tilstøtende histoner, og L1L2 -stedet dannet av L1- og L2 -løkkene. Saltledd og hydrogenbinding mellom både sidekjede grunnleggende og hydroksylgrupper og hovedkjede amider med DNA-ryggradsfosfatene utgjør hoveddelen av interaksjoner med DNA. Dette er viktig, gitt at allestedsnærværende fordeling av nukleosomer langs genomer krever at det er en ikke-sekvens-spesifikk DNA-bindende faktor. Selv om nukleosomer har en tendens til å foretrekke noen DNA-sekvenser fremfor andre, er de i stand til å binde praktisk talt til enhver sekvens, noe som antas å skyldes fleksibiliteten i dannelsen av disse vannmedierte interaksjonene. I tillegg utføres ikke-polare interaksjoner mellom proteinsidekjeder og deoksyribosegruppene, og en argininsidekjede interkalerer inn i DNA-mindre spor på alle 14 steder der den vender mot oktameroverflaten. Fordelingen og styrken til DNA-bindingssteder om oktameroverflaten forvrenger DNA i nukleosomkjernen. DNA er ujevnt bøyd og inneholder også vridningsdefekter. Vridningen av fritt B-form-DNA i oppløsning er 10,5 bp per tur. Imidlertid er den totale vridningen av nukleosomalt DNA bare 10,2 bp per sving, som varierer fra en verdi på 9,4 til 10,9 bp per sving.
Histone haledomener
Histonhale -forlengelsene utgjør opptil 30% av massen av histoner, men er ikke synlige i krystallstrukturene i nukleosomer på grunn av deres høye iboende fleksibilitet, og har blitt antatt å være stort sett ustrukturerte. De N-terminale halene til histonene H3 og H2B passerer gjennom en kanal dannet av de mindre sporene i de to DNA-strengene, som stikker ut fra DNA hvert 20. bp. Den N-terminale halen til histon H4 har derimot et område med svært basiske aminosyrer (16-25), som i krystallstrukturen danner en interaksjon med det svært sure overflateområdet til en H2A-H2B-dimer av et annet nukleosom, som potensielt er relevant for strukturen av høyere orden av nukleosomer. Denne interaksjonen antas også å forekomme under fysiologiske forhold, og antyder at acetylering av H4-halen forvrider strukturen av høyere orden av kromatin.
Høyere ordrestruktur
Organiseringen av DNA som nukleosomet oppnår kan ikke fullt ut forklare emballasjen til DNA observert i cellekjernen. Ytterligere komprimering av kromatin inn i cellekjernen er nødvendig, men det er ennå ikke godt forstått. Den nåværende forståelsen er at repeterende nukleosomer med mellomliggende "linker" DNA danner en 10-nm-fiber , beskrevet som "perler på en streng", og har et pakningsforhold på omtrent fem til ti. En kjede av nukleosomer kan arrangeres i en 30 nm fiber , en komprimert struktur med et pakningsforhold på ~ 50 og hvis dannelse er avhengig av tilstedeværelsen av H1 -histonet .
En krystallstruktur av et tetranukleosom har blitt presentert og brukt til å bygge opp en foreslått struktur av 30 nm fiber som en to-start helix. Det er fortsatt en viss påstand om denne modellen, siden den er inkompatibel med nylige elektronmikroskopidata . Utover dette er strukturen til kromatin dårlig forstått, men det er klassisk antydet at 30 nm fiberen er ordnet i sløyfer langs et sentralt protein -stillas for å danne transkripsjonelt aktivt eukromatin . Ytterligere komprimering fører til transkripsjonelt inaktivt heterokromatin .
Dynamikk
Selv om nukleosomet er et veldig stabilt protein-DNA-kompleks, er det ikke statisk og har vist seg å gjennomgå en rekke forskjellige strukturelle omarrangementer, inkludert nukleosomglidning og eksponering av DNA-stedet. Avhengig av konteksten kan nukleosomer hemme eller lette transkripsjonsfaktorbinding. Nukleosomposisjoner styres av tre hovedbidrag: For det første avhenger histonoktamerens iboende bindingsaffinitet av DNA -sekvensen. For det andre kan nukleosomet fortrenges eller rekrutteres ved konkurrerende eller samarbeidende binding av andre proteinfaktorer. For det tredje kan nukleosomet aktivt translokeres av ATP-avhengige ombyggingskomplekser.
Nukleosom glidende
Arbeid utført i Bradbury -laboratoriet viste at nukleosomer rekonstituert på 5S DNA -posisjoneringssekvensen var i stand til å reposisjonere seg translasjonelt på tilstøtende sekvenser når de ble inkubert termisk. Senere arbeid viste at denne reposisjonen ikke krevde forstyrrelse av histonoktameren, men var i samsvar med at nukleosomer kunne "gli" langs DNA i cis . I 2008 ble det videre avslørt at CTCF -bindingssteder fungerer som nukleosomposisjonsankre, slik at flere flankerende nukleosomer lett kan identifiseres når de brukes til å justere forskjellige genomiske signaler. Selv om nukleosomer er iboende mobile, har eukaryoter utviklet en stor familie av ATP-avhengige kromatin-remodelleringsenzymer for å endre kromatinstrukturen, hvorav mange gjør det via nukleosomglidning. I 2012 har Beena Pillais laboratorium demonstrert at nukleosomglidning er en av de mulige mekanismene for vevsspesifikk uttrykk for gener i stor skala. Arbeidet viser at transkripsjonens startsted for gener uttrykt i et bestemt vev, er nukleosomutarmet, mens det samme settet med gener i annet vev der de ikke uttrykkes, er nukleosombundet.
Eksponering av DNA -stedet
Arbeid fra Widom -laboratoriet har vist at nukleosomalt DNA er i likevekt mellom en innpakket og uinnpakket tilstand. Målinger av disse hastighetene ved bruk av tidsoppløst FRET avslørte at DNA i nukleosomet forblir fullstendig innpakket i bare 250 ms før det pakkes ut i 10-50 ms og deretter raskt ompakkes. Dette innebærer at DNA ikke trenger å være aktivt dissosiert fra nukleosomet, men at det er en betydelig brøkdel av tiden det er fullt tilgjengelig. Dette kan faktisk utvides til observasjonen av at innføring av en DNA-bindende sekvens i nukleosomet øker tilgjengeligheten til tilstøtende DNA-områder når den er bundet. Denne tilbøyeligheten for DNA i nukleosomet til å "puste" har viktige funksjonelle konsekvenser for alle DNA-bindende proteiner som opererer i et kromatinmiljø. Spesielt spiller den dynamiske pusten til nukleosomer en viktig rolle i å begrense fremskrittet av RNA -polymerase II under transkripsjonsforlengelse.
Nukleosomfri region
Promotører av aktive gener har nukleosomfrie regioner (NFR). Dette gir mulighet for promotor -DNA -tilgang til forskjellige proteiner, for eksempel transkripsjonsfaktorer. Nukleosomfri region spenner vanligvis over 200 nukleotider i S. cerevisae Godt posisjonerte nukleosomer danner grenser for NFR. Disse nukleosomene kalles +1-nukleosom og −1-nukleosom og ligger på kanoniske avstander henholdsvis nedstrøms og oppstrøms fra transkripsjonsstartstedet. +1-nukleosom og flere nedstrøms nukleosomer har også en tendens til å innlemme H2A.Z histonvariant.
Modulerende nukleosomstruktur
Eukaryote genomer er allestedsnærværende assosiert med kromatin; imidlertid må cellene romlig og tidsmessig regulere spesifikke lokaliteter uavhengig av bulk -kromatin. For å oppnå det høye kontrollnivået som kreves for å koordinere kjernefysiske prosesser som DNA-replikasjon, reparasjon og transkripsjon, har celler utviklet en rekke metoder for lokalt og spesifikt å modulere kromatinstruktur og funksjon. Dette kan innebære kovalent modifikasjon av histoner, inkorporering av histonvarianter og ikke-kovalent remodellering av ATP-avhengige remodelleringsenzymer.
Histone post-translasjonelle modifikasjoner
Siden de ble oppdaget på midten av 1960-tallet, har histonendringer blitt spådd å påvirke transkripsjonen. Det faktum at de fleste av de tidlige posttranslasjonelle modifikasjonene som ble funnet var konsentrert i haleforlengelsene som stikker ut fra nukleosomkjernen, fører til to hovedteorier om mekanismen for histonmodifisering. Den første av teoriene antydet at de kan påvirke elektrostatiske interaksjoner mellom histonhalene og DNA for å "løsne" kromatinstrukturen. Senere ble det foreslått at kombinasjoner av disse modifikasjonene kan skape bindende epitoper for rekruttering av andre proteiner. Siden det nylig er funnet flere modifikasjoner i de strukturerte områdene i histoner, har det blitt fremmet at disse modifikasjonene kan påvirke histon-DNA og histon-histon-interaksjoner i nukleosomkjernen. Modifikasjoner (for eksempel acetylering eller fosforylering) som senker ladningen til den globulære histonkjernen er spådd å "løsne" kjerne-DNA-forening; effekten av effekten avhenger av plasseringen av modifikasjonen i kjernen. Noen modifikasjoner har vist seg å være korrelert med gendemping; andre ser ut til å være korrelert med genaktivering. Vanlige modifikasjoner inkluderer acetylering , metylering eller ubiquitination av lysin ; metylering av arginin ; og fosforylering av serin . Informasjonen som er lagret på denne måten regnes som epigenetisk , siden den ikke er kodet i DNA, men fortsatt arves til datterceller. Opprettholdelse av en undertrykt eller aktivert status for et gen er ofte nødvendig for celledifferensiering .
Histonvarianter
Selv om histoner er bemerkelsesverdig bevart gjennom evolusjonen, har flere variantformer blitt identifisert. Denne diversifiseringen av histonfunksjonen er begrenset til H2A og H3, og H2B og H4 er stort sett invariante. H2A kan erstattes av H2AZ (noe som fører til redusert nukleosomstabilitet) eller H2AX (som er assosiert med DNA -reparasjon og T -celledifferensiering ), mens de inaktive X -kromosomene hos pattedyr er beriket med makroH2A. H3 kan erstattes av H3.3 (som korrelerer med aktiverte gener og regulatoriske elementer) og i sentromerer erstattes H3 av CENPA .
ATP-avhengig nukleosom-ombygging
En rekke forskjellige reaksjoner er assosiert med begrepet ATP-avhengig kromatin-ombygging . Remodelleringsenzymer har vist seg å skyve nukleosomer langs DNA, forstyrre histon-DNA-kontakter i den grad at de destabiliserer H2A/H2B-dimeren og genererer negativ superhelisk torsjon i DNA og kromatin. Nylig har Swr1 -remodelleringsenzymet vist seg å introdusere varianten histon H2A.Z i nukleosomer. For tiden er det ikke klart om alle disse representerer forskjellige reaksjoner eller bare alternative utfall av en felles mekanisme. Det som deles mellom alle, og faktisk kjennetegnet ved ATP-avhengig kromatin-ombygging, er at de alle resulterer i endret DNA-tilgjengelighet.
Studier som ser på genaktivering in vivo og mer overraskende, ombygging in vitro har avslørt at kromatin-ombyggingshendelser og transkripsjonsfaktorbinding er syklisk og periodisk. Selv om konsekvensene av dette for reaksjonsmekanismen ved kromatin -ombygging ikke er kjent, kan systemets dynamiske natur tillate det å reagere raskere på ytre stimuli. En fersk studie indikerer at nukleosomposisjoner endres betydelig under musens embryonale stamcelleutvikling, og disse endringene er knyttet til binding av utviklingstranskripsjonsfaktorer.
Dynamisk nukleosom -ombygging på tvers av gjærgenomet
Studier i 2007 har katalogisert nukleosomposisjoner i gjær og vist at nukleosomer er utarmet i promoterregioner og replikasjonens opprinnelse . Omtrent 80% av gjærgenomet ser ut til å være dekket av nukleosomer, og mønsteret for nukleosomposisjonering angår tydelig DNA -regioner som regulerer transkripsjon , regioner som transkriberes og regioner som starter DNA -replikasjon. Nylig undersøkte en ny studie dynamiske endringer i nukleosomposisjonering under en global transkripsjonell omprogrammeringshendelse for å belyse effektene på nukleosomforskyvning under genomgående transkripsjonelle endringer i gjær ( Saccharomyces cerevisiae ). Resultatene antydet at nukleosomer som var lokalisert til promoterregionene blir fortrengt som respons på stress (som varmesjokk ). I tillegg tilsvarte fjerning av nukleosomer vanligvis transkripsjonell aktivering og erstatning av nukleosomer tilsvarte vanligvis transkripsjonell undertrykkelse, antagelig fordi transkripsjonsfaktorbindingssteder ble mer eller mindre tilgjengelige, henholdsvis. Generelt ble bare ett eller to nukleosomer flyttet til promotoren for å utføre disse transkripsjonelle endringene. Selv i kromosomale regioner som ikke var assosiert med transkripsjonelle endringer, ble det imidlertid observert omplassering av nukleosomer, noe som tyder på at dekning og avdekking av transkripsjonelt DNA ikke nødvendigvis gir en transkripsjonell hendelse. Etter transkripsjon må rDNA -regionen beskyttes mot enhver skade, det antydet at HMGB -proteiner spiller en viktig rolle i å beskytte den nukleosomfrie regionen.
Nukleosommontering in vitro
Nukleosomer kan samles in vitro ved enten å bruke rensede native eller rekombinante histoner. En standard teknikk for lasting av DNA rundt histonene innebærer bruk av saltdialyse . En reaksjon bestående av histonoktamerene og en naken DNA -mal kan inkuberes sammen ved en saltkonsentrasjon på 2 M. Ved jevnt å redusere saltkonsentrasjonen vil DNA ekvilibrere til en posisjon hvor det er viklet rundt histonoktamerene og danne nukleosomer. Under passende forhold tillater denne rekonstitueringsprosessen at nukleosomposisjoneringsaffiniteten til en gitt sekvens kan kartlegges eksperimentelt.
Disulfid -tverrbundne nukleosomkjernepartikler
En nylig fremgang i produksjonen av nukleosomkjernepartikler med forbedret stabilitet involverer stedsspesifikke disulfid- tverrbindinger. To forskjellige tverrbindinger kan innføres i nukleosomkjernepartikkelen. Den første tverrbinder de to kopiene av H2A via et introdusert cystein (N38C), noe som resulterer i histonoktamer som er stabilt mot tap av H2A/H2B -dimer under nukleosomrekonstituering. En andre tverrbinding kan innføres mellom den H3 N-terminale histonhale og nukleosom-DNA-ender via et inkorporert konvertibelt nukleotid. DNA-histonoktamer-tverrbindingen stabiliserer nukleosomkjernepartikkelen mot DNA-dissosiasjon ved meget lave partikkelkonsentrasjoner og ved forhøyede saltkonsentrasjoner.
Nukleosommontering in vivo
Nukleosomer er den grunnleggende pakningsenheten til DNA bygget av histonproteiner som DNA er spolet rundt. De fungerer som et stillas for dannelse av kromatinstruktur av høyere orden, så vel som for et lag med regulatorisk kontroll av genuttrykk. Nukleosomer blir raskt satt sammen på nysyntetisert DNA bak replikasjonsgaffelen.
H3 og H4
Histoner H3 og H4 fra demonterte gamle nukleosomer oppbevares i nærheten og fordeles tilfeldig på det nylig syntetiserte DNA. De er satt sammen av kromatinsammensetningsfaktor-1 (CAF-1) -komplekset, som består av tre underenheter (p150, p60 og p48). Nysyntetisert H3 og H4 settes sammen av replikasjonskoblingsmonteringsfaktoren (RCAF). RCAF inneholder underenheten Asf1, som binder seg til nysyntetiserte H3- og H4 -proteiner. De gamle H3- og H4 -proteinene beholder sine kjemiske modifikasjoner som bidrar til at den epigenetiske signaturen går ned. De nysyntetiserte H3- og H4 -proteinene acetyleres gradvis ved forskjellige lysinrester som en del av kromatinmodningsprosessen. Det antas også at de gamle H3- og H4 -proteinene i de nye nukleosomene rekrutterer histonmodifiserende enzymer som markerer de nye histonene, noe som bidrar til epigenetisk minne.
H2A og H2B
I motsetning til gamle H3 og H4 frigjøres og nedbrytes de gamle H2A- og H2B -histonproteinene; derfor er nymonterte H2A- og H2B -proteiner inkorporert i nye nukleosomer. H2A og H2B settes sammen til dimerer som deretter lastes inn på nukleosomer av nukleosomsammensetningsprotein-1 (NAP-1) som også hjelper til med glidning av nukleosomer. Nukleosomene er også fordelt på ATP-avhengige nukleosom-ombyggingskomplekser som inneholder enzymer som Isw1 Ino80 og Chd1, og blir deretter satt sammen til en struktur av høyere orden.
Galleri
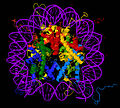
Krystallstrukturen til nukleosomkjernepartikkelen ( PDB : 1EQZ ) - forskjellige visninger som viser detaljer om histonbretting og organisering. Histoner H2A , H2B , H3 , H4 og DNA er farget.
Se også
Referanser
Eksterne linker
- MBInfo - Hva er nukleosomer
- Nukleosomer på Richmond Lab -nettstedet
- Proteopedia nucleosomes
- Nukleosom ved PDB
- Dynamisk ombygging av individuelle nukleosomer over et eukaryotisk genom som svar på transkripsjonell forstyrrelse
- Nukleosomposisjonsdata og verktøy online (kommentert liste, oppdateres kontinuerlig)
- Histonproteinstruktur
- HistoneDB 2.0 - Database over histoner og varianter ved NCBI