Sekvensjustering - Sequence alignment
I bioinformatikk er en sekvensjustering en måte å arrangere sekvensene av DNA , RNA eller protein for å identifisere områder med likhet som kan være en konsekvens av funksjonelle, strukturelle eller evolusjonære forhold mellom sekvensene. Justerte sekvenser av nukleotid- eller aminosyrerester er vanligvis representert som rader i en matrise . Det settes inn hull mellom restene slik at identiske eller lignende tegn er justert i påfølgende kolonner. Sekvensjusteringer brukes også for ikke-biologiske sekvenser, for eksempel beregning av avstandskostnaden mellom strenger på et naturlig språk eller i økonomiske data.

Sekvenser er aminosyrene for restene 120-180 av proteinene. Rester som er konservert på tvers av alle sekvenser er markert med grått. Under proteinsekvensene er en nøkkel som angir konservert sekvens (*), konservative mutasjoner (:), semikonservative mutasjoner (.) Og ikke-konservative mutasjoner ().
Tolkning
Hvis to sekvenser i en justering deler en felles stamfar, kan mismatches tolkes som punktmutasjoner og hull som indeler (det vil si innsetting eller slettingmutasjoner) introdusert i en eller begge slektninger i tiden siden de avviker fra hverandre. I sekvensjusteringer av proteiner kan graden av likhet mellom aminosyrer som inntar en bestemt posisjon i sekvensen tolkes som et grovt mål på hvor konservert et bestemt område eller sekvensmotiv er blant slektninger. Fraværet av substitusjoner, eller tilstedeværelsen av bare veldig konservative substitusjoner (det vil si substitusjon av aminosyrer hvis sidekjeder har lignende biokjemiske egenskaper) i en bestemt region i sekvensen, antyder at denne regionen har strukturell eller funksjonell betydning. Selv om DNA- og RNA -nukleotidbaser ligner mer på hverandre enn aminosyrer, kan bevaring av basepar indikere en lignende funksjonell eller strukturell rolle.
Justeringsmetoder
Svært korte eller veldig like sekvenser kan justeres for hånd. De fleste interessante problemene krever imidlertid justering av lange, svært variable eller ekstremt mange sekvenser som ikke kan justeres utelukkende ved menneskelig innsats. I stedet brukes menneskelig kunnskap i konstruksjon av algoritmer for å produsere sekvensjusteringer av høy kvalitet, og noen ganger i å justere de endelige resultatene for å gjenspeile mønstre som er vanskelig å representere algoritmisk (spesielt når det gjelder nukleotidsekvenser). Beregningsmetoder for sekvensjustering faller vanligvis i to kategorier: globale justeringer og lokale justeringer . Beregning av en global justering er en form for global optimalisering som "tvinger" justeringen til å spenne over hele lengden på alle spørresekvenser. Derimot identifiserer lokale justeringer regioner med likhet i lange sekvenser som ofte er vidt divergerende totalt sett. Lokale justeringer er ofte å foretrekke, men kan være vanskeligere å beregne på grunn av den ekstra utfordringen med å identifisere likhetsregionene. En rekke beregningsalgoritmer har blitt brukt på sekvensjusteringsproblemet. Disse inkluderer langsomme, men formelt korrekte metoder som dynamisk programmering . Disse inkluderer også effektive, heuristiske algoritmer eller sannsynlighetsmetoder designet for storstilt databasesøk, som ikke garanterer å finne de beste treffene.
Representasjoner
Justeringer er ofte representert både grafisk og i tekstformat. I nesten alle sekvensjusteringsrepresentasjoner skrives sekvenser i rader ordnet slik at justerte rester vises i påfølgende kolonner. I tekstformater er justerte kolonner som inneholder identiske eller lignende tegn angitt med et system med bevaringssymboler. Som på bildet ovenfor, brukes en stjerne eller et rørsymbol for å vise identitet mellom to kolonner; andre mindre vanlige symboler inkluderer et kolon for konservative substitusjoner og en periode for semikonservative substitusjoner. Mange sekvensvisualiseringsprogrammer bruker også farger for å vise informasjon om egenskapene til de enkelte sekvenselementene; i DNA- og RNA -sekvenser tilsvarer dette å tildele hvert nukleotid sin egen farge. I proteinjusteringer, som den på bildet ovenfor, brukes ofte farge for å indikere aminosyreegenskaper for å hjelpe til med å bedømme bevaringen av en gitt aminosyresubstitusjon. For flere sekvenser er den siste raden i hver kolonne ofte konsensussekvensen som er bestemt av justeringen; konsensus -sekvensen er også ofte representert i grafisk format med en sekvenslogo der størrelsen på hvert nukleotid eller aminosyrebokstav tilsvarer dens grad av bevaring.
Sekvensjusteringer kan lagres i en lang rekke tekstbaserte filformater, hvorav mange opprinnelig ble utviklet i forbindelse med et spesifikt justeringsprogram eller en implementering. De fleste nettbaserte verktøy tillater et begrenset antall input- og output-formater, for eksempel FASTA-format og GenBank- format, og utdataene kan ikke enkelt redigeres. Flere konverteringsprogrammer som gir grafiske og/eller kommandolinjegrensesnitt er tilgjengelige, for eksempel READSEQ og EMBOSS . Det er også flere programmeringspakker som gir denne konverteringsfunksjonaliteten, for eksempel BioPython , BioRuby og BioPerl . De SAM / BAM-filer bruke CIGAR (Compact Idiosyncratic gapped Alignment Rapporter) strengformat for å representere en justering av en sekvens til en referanse ved koding av en sekvens av hendelser (f.eks kamp / mismatch, insersjoner, delesjoner).
CIGAR -format
Ref. : GTCGTAGAATA
Les : CACGTAG — TA
CIGAR: 2S5M2D2M hvor:
2S = 2 myk klipping (kan være feil eller en lesning som er lengre enn den matchede sekvensen)
5M = 5 treff eller feil
2D = 2 slettinger
2M = 2 treff eller feil samsvar
Det originale CIGAR -formatet fra programmet for frigjøring av justeringer skilte ikke mellom feil samsvar eller samsvar med M -tegnet.
SAMv1 -spesifikasjonsdokumentet definerer nyere CIGAR -koder. I de fleste tilfeller er det foretrukket å bruke '=' og 'X' -tegnene for å angi treff eller uoverensstemmelser i stedet for det eldre 'M' -tegnet, som er tvetydig.
| CIGAR -kode | BAM Heltall | Beskrivelse | Forbruker spørring | Bruker referanse |
| M | 0 | justeringskamp (kan være en sekvensmatch eller feil samsvar) | ja | ja |
| Jeg | 1 | innsetting til referansen | ja | Nei |
| D | 2 | sletting fra referansen | Nei | ja |
| N | 3 | hoppet over regionen fra referansen | Nei | ja |
| S | 4 | myk klipping (klippte sekvenser tilstede i SEQ) | ja | Nei |
| H | 5 | hardt klipping (klippte sekvenser IKKE tilstede i SEQ) | Nei | Nei |
| P | 6 | polstring (stille sletting fra polstret referanse) | Nei | Nei |
| = | 7 | sekvensmatch | ja | ja |
| X | 8 | sekvensmangel | ja | ja |
- "Forbruker spørring" og "forbruker referanse" indikerer om CIGAR -operasjonen får justeringen til å gå henholdsvis spørresekvensen og referansesekvensen.
- H kan bare være tilstede som den første og/eller siste operasjonen.
- S kan bare ha H -operasjoner mellom dem og endene på CIGAR -strengen.
- For mRNA-til-genom-justering representerer en N-operasjon et intron. For andre typer justeringer er tolkningen av N ikke definert.
- Summen av lengder på M/I/S/=/X -operasjonene skal være lik lengden på SEQ
Globale og lokale justeringer
Globale justeringer, som prøver å justere hver rest i hver sekvens, er mest nyttige når sekvensene i spørresettet er like og omtrent like store. (Dette betyr ikke at globale justeringer ikke kan starte og/eller ende i hull.) En generell global justeringsteknikk er Needleman - Wunsch -algoritmen , som er basert på dynamisk programmering. Lokale justeringer er mer nyttige for forskjellige sekvenser som mistenkes å inneholde områder med likhet eller lignende sekvensmotiver innenfor deres større sekvenskontekst. Den Smith-Waterman algoritmen er en generell lokal justering metode basert på den samme dynamisk programmering ordningen, men med flere valg å starte og avslutte på ethvert sted.
Hybridmetoder, kjent som semi-global eller "glocal" (kort for glo bal-lo cal ) metoder, søker etter best mulig delvis justering av de to sekvensene (med andre ord en kombinasjon av en eller begge starter og en eller begge deler ender er angitt å være justert). Dette kan være spesielt nyttig når nedstrøms delen av en sekvens overlapper med oppstrøms delen av den andre sekvensen. I dette tilfellet er verken global eller lokal justering helt hensiktsmessig: en global justering vil forsøke å tvinge justeringen til å strekke seg utover overlappingsområdet, mens en lokal justering kanskje ikke fullt ut dekker overlappingsområdet. Et annet tilfelle der semi-global justering er nyttig er når den ene sekvensen er kort (for eksempel en gensekvens) og den andre er veldig lang (for eksempel en kromosomsekvens). I så fall bør den korte sekvensen være globalt (helt) justert, men bare en lokal (delvis) justering er ønsket for den lange sekvensen.
Rask utvidelse av genetiske data utfordrer hastigheten på nåværende algoritmer for justering av DNA -sekvens. Viktige behov for en effektiv og nøyaktig metode for DNA -variantoppdagelse krever innovative tilnærminger for parallellbehandling i sanntid. Optiske databehandlingsmetoder er blitt foreslått som lovende alternativer til de nåværende elektriske implementeringene, men anvendeligheten gjenstår å teste [1] .
Justering i par
Parvisvise sekvensjusteringsmetoder brukes til å finne de best matchende stykkevise (lokale eller globale) justeringene av to spørresekvenser. Justeringer i par kan bare brukes mellom to sekvenser om gangen, men de er effektive å beregne og brukes ofte for metoder som ikke krever ekstrem presisjon (for eksempel å søke i en database etter sekvenser med høy likhet med et søk). De tre primære metodene for å produsere parvise justeringer er dot-matrix-metoder, dynamisk programmering og ordmetoder; Imidlertid kan flere sekvensjusteringsteknikker også justere sekvenspar. Selv om hver metode har sine individuelle styrker og svakheter, har alle tre parvise metodene problemer med svært repeterende sekvenser med lavt informasjonsinnhold - spesielt der antall repetisjoner er forskjellige i de to sekvensene som skal justeres.
Maksimal unik match
En måte å kvantifisere nytten av en gitt parvis justering er ' maksimal unik match ' (MUM), eller den lengste undersekvensen som oppstår i begge spørresekvensene. Lengre MUM -sekvenser gjenspeiler vanligvis tettere slektskap. i flersekvensjustering av genomer i beregningsbiologi . Identifisering av MUMs og andre potensielle ankre er det første trinnet i større justeringssystemer som MUMmer . Anker er områdene mellom to genomer der de er svært like. For å forstå hva en mamma er, kan vi bryte ned hvert ord i akronymet. Match innebærer at delstrengen forekommer i begge sekvensene som skal justeres. Unikt betyr at delstrengen bare forekommer én gang i hver sekvens. Til slutt angir maksimal at delstrengen ikke er en del av en annen større streng som oppfyller begge tidligere krav. Ideen bak dette er at lange sekvenser som samsvarer nøyaktig og som bare forekommer én gang i hvert genom nesten er en del av den globale justeringen.
Mer presist:
"Gitt to genomer A og B, er maksimal Unique Match (MUM) delstreng en vanlig delstreng på A og B lengre enn en spesifisert minimumslengde d (som standard d = 20) slik at
- den er maksimal, det vil si at den ikke kan forlenges i begge ender uten å pådra seg en mismatch; og
- det er unikt i begge sekvensene "
Punktmatrisemetoder

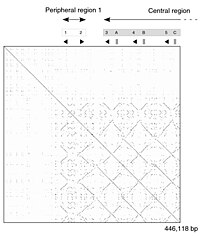
Et DNA dot plott av en human zinkfinger transkripsjonsfaktor (GenBank ID NM_002383), som viser regional egenlikhet . Hoveddiagonalen representerer sekvensens justering med seg selv; linjer utenfor hoveddiagonalen representerer lignende eller repeterende mønstre i sekvensen. Dette er et typisk eksempel på en gjentagelsesplott .
|
Punktmatrisemetoden, som implisitt produserer en familie med justeringer for individuelle sekvensområder, er kvalitativ og konseptuelt enkel, men tidkrevende å analysere i stor skala. I fravær av støy kan det være enkelt å visuelt identifisere visse sekvensfunksjoner-for eksempel innsetting, sletting, gjentakelse eller omvendt gjentagelse- fra et punktmatriseplot. For å konstruere et prikk-matriksplott , skrives de to sekvensene langs den øverste raden og venstre kolonne i en todimensjonal matrise, og en prikk plasseres på et hvilket som helst punkt der tegnene i de aktuelle kolonnene stemmer overens-dette er et typisk gjentagelsesplott . Noen implementeringer varierer størrelsen eller intensiteten til prikken, avhengig av graden av likhet mellom de to karakterene, for å imøtekomme konservative substitusjoner. Prikkplottene for svært nært beslektede sekvenser vil vises som en enkelt linje langs matrisens hoveddiagonal .
Problemer med prikkplott som informasjonsvisningsteknikk inkluderer: støy, mangel på klarhet, ikke-intuitivitet, vanskeligheter med å trekke ut statistikk for kampoppsummering og kampposisjoner på de to sekvensene. Det er også mye bortkastet plass der kampdataene iboende dupliseres på tvers av diagonalen, og det meste av plottets faktiske areal blir tatt opp av enten tomt rom eller støy, og til slutt er prikkdiagrammer begrenset til to sekvenser. Ingen av disse begrensningene gjelder for Miropeats justeringsdiagrammer, men de har sine egne spesielle feil.
Prikkplott kan også brukes til å vurdere repetitivitet i en enkelt sekvens. En sekvens kan plottes mot seg selv, og regioner som har betydelige likheter vil vises som linjer utenfor hoveddiagonalen. Denne effekten kan oppstå når et protein består av flere lignende strukturelle domener .
Dynamisk programmering
Teknikken for dynamisk programmering kan brukes for å produsere globale justeringer via Needleman-Wunsch-algoritmen , og lokale justeringer via Smith-Waterman-algoritmen . Ved typisk bruk bruker proteinjusteringer en substitusjonsmatrise for å tildele poengsummer til aminosyrematcher eller feilpasninger, og en gapstraff for å matche en aminosyre i den ene sekvensen til et gap i den andre. DNA- og RNA -justeringer kan bruke en scoringsmatrise, men tildeler i praksis ofte bare en positiv matchscore, en negativ mismatch -score og en negativ gap -straff. (I standard dynamisk programmering, at resultatet av hver aminosyre posisjon er uavhengig av identiteten til sine naboer, og derfor basisstablings effekter blir ikke tatt i betraktning. Det er imidlertid mulig å ta hensyn til slike effekter ved å endre den algoritmen.) En vanlig utvidelse til standard lineære gapkostnader, er bruken av to forskjellige gapstraffer for å åpne et gap og for å utvide et gap. Typisk er førstnevnte mye større enn sistnevnte, f.eks. -10 for åpning åpent og -2 for gapforlengelse. Dermed blir antallet hull i en justering vanligvis redusert og rester og hull holdes sammen, noe som vanligvis gir mer biologisk mening. Gotoh -algoritmen implementerer affine gap -kostnader ved å bruke tre matriser.
Dynamisk programmering kan være nyttig for å tilpasse nukleotid til proteinsekvenser, en oppgave komplisert av behovet for å ta hensyn til rammeskiftmutasjoner (vanligvis innsetting eller sletting). Rammesøkingsmetoden produserer en rekke globale eller lokale parvise justeringer mellom en spørringsnukleotidsekvens og et søkesett med proteinsekvenser, eller omvendt. Dens evne til å evaluere rammeskift kompensert av et vilkårlig antall nukleotider gjør metoden nyttig for sekvenser som inneholder et stort antall indeler, noe som kan være svært vanskelig å justere med mer effektive heuristiske metoder. I praksis krever metoden store datamengder eller et system hvis arkitektur er spesialisert for dynamisk programmering. De BLAST og preging suiter tilbyr grunnleggende verktøy for å lage oversatte justeringer (selv om noen av disse nærmer dra nytte av bivirkninger av sekvens søker evner av verktøy). Mer generelle metoder er tilgjengelige fra åpen kildekode-programvare som GeneWise .
Den dynamiske programmeringsmetoden finner garantert en optimal justering gitt en bestemt poengsumfunksjon; Imidlertid er det ofte en empirisk fremfor en teoretisk sak å identifisere en god poengsumfunksjon. Selv om dynamisk programmering kan utvides til mer enn to sekvenser, er det uoverkommelig tregt for et stort antall sekvenser eller ekstremt lange sekvenser.
Ordmetoder
Ordmetoder , også kjent som k -tuple -metoder, er heuristiske metoder som ikke garantert finner en optimal justeringsløsning, men er betydelig mer effektive enn dynamisk programmering. Disse metodene er spesielt nyttige i store databasesøk der det er forstått at en stor andel av kandidatsekvensene i det vesentlige ikke vil ha noen signifikant samsvar med forespørselssekvensen. Ordmetoder er best kjent for implementering i databasesøkverktøyene FASTA og BLAST -familien. Ordmetoder identifiserer en serie korte, ikke -overlappende undersekvenser ("ord") i spørresekvensen som deretter blir matchet med kandidatdatabasesekvenser. De relative posisjonene til ordet i de to sekvensene som blir sammenlignet, trekkes fra for å oppnå en forskyvning; dette vil indikere et område for justering hvis flere forskjellige ord gir samme forskyvning. Bare hvis denne regionen blir oppdaget, bruker disse metodene mer sensitive justeringskriterier; Dermed elimineres mange unødvendige sammenligninger med sekvenser uten nevneverdig likhet.
I FASTA -metoden definerer brukeren en verdi k som skal brukes som ordlengden for å søke i databasen. Metoden er tregere, men mer sensitiv ved lavere verdier av k , som også er foretrukket for søk som involverer en veldig kort spørresekvens. BLAST -familien av søkemetoder gir en rekke algoritmer optimalisert for bestemte typer søk, for eksempel å søke etter fjernt beslektede sekvensmatcher. BLAST ble utviklet for å gi et raskere alternativ til FASTA uten å ofre mye nøyaktighet; i likhet med FASTA, bruker BLAST et ordsøk med lengde k , men evaluerer bare de mest betydningsfulle ordmatchene, i stedet for hvert ord som FASTA gjør. De fleste BLAST -implementeringer bruker en fast standard ordlengde som er optimalisert for spørringen og databasetypen, og som bare endres under spesielle omstendigheter, for eksempel når du søker med repeterende eller svært korte spørresekvenser. Implementeringene finnes via en rekke nettportaler, for eksempel EMBL FASTA og NCBI BLAST .
Justering av flere sekvenser

Justering av flere sekvenser er en forlengelse av parvis justering for å inkludere mer enn to sekvenser om gangen. Flere justeringsmetoder prøver å justere alle sekvensene i et gitt spørresett. Flere justeringer brukes ofte for å identifisere bevarte sekvensområder på tvers av en gruppe sekvenser som antas å være evolusjonært relaterte. Slike konserverte sekvensmotiver kan brukes i forbindelse med strukturell og mekanistisk informasjon for å lokalisere de katalytiske aktive setene til enzymer . Justeringer brukes også til å hjelpe til med å etablere evolusjonære forhold ved å bygge fylogenetiske trær . Flere sekvensjusteringer er beregningsmessig vanskelige å produsere, og de fleste formuleringer av problemet fører til NP-komplette kombinatoriske optimaliseringsproblemer. Likevel har nytten av disse justeringene i bioinformatikk ført til utviklingen av en rekke metoder som er egnet for å tilpasse tre eller flere sekvenser.
Dynamisk programmering
Teknikken for dynamisk programmering er teoretisk anvendelig for et hvilket som helst antall sekvenser; Fordi det er beregningsmessig dyrt både i tid og minne , brukes det imidlertid sjelden i mer enn tre eller fire sekvenser i sin mest grunnleggende form. Denne metoden krever konstruksjon av den n -dimensjonale tilsvarer sekvensen matrise dannet av to sekvenser, hvor n er antallet sekvenser i spørringen. Standard dynamisk programmering brukes først på alle par forespørselssekvenser, og deretter fylles "justeringsrommet" ved å vurdere mulige treff eller hull i mellomposisjoner, og til slutt konstruere en justering i hovedsak mellom hver to-sekvens justering. Selv om denne teknikken er beregningsmessig dyr, er garantien for en global optimal løsning nyttig i tilfeller der bare noen få sekvenser må justeres nøyaktig. En metode for å redusere beregningskravene til dynamisk programmering, som er avhengig av "summen av par" -funksjonen , er implementert i MSA -programvarepakken.
Progressive metoder
Progressive, hierarkiske eller tremetoder genererer en justering av flere sekvenser ved først å tilpasse de mest lignende sekvensene og deretter legge til påfølgende mindre beslektede sekvenser eller grupper til justeringen til hele spørresettet er inkorporert i løsningen. Det innledende treet som beskriver sekvensrelatiteten er basert på parvise sammenligninger som kan inkludere heuristiske parvise justeringsmetoder som ligner på FASTA . Progressive justeringsresultater er avhengig av valget av "mest relaterte" sekvenser og kan dermed være følsomme for unøyaktigheter i de første parvise justeringene. De fleste progressive flere sekvensjusteringsmetodene veier i tillegg sekvensene i spørresettet i henhold til deres slektskap, noe som reduserer sannsynligheten for å gjøre et dårlig valg av innledende sekvenser og dermed forbedrer justeringsnøyaktigheten.
Mange varianter av den progressive Clustal -implementeringen brukes til justering av flere sekvenser, fylogenetisk trekonstruksjon og som innspill til forutsigelse av proteinstruktur . En tregere, men mer nøyaktig variant av den progressive metoden er kjent som T-Coffee .
Iterative metoder
Iterative metoder prøver å forbedre den store avhengigheten av nøyaktigheten av de første parvise justeringene, som er det svake punktet for de progressive metodene. Iterative metoder optimaliserer en objektiv funksjon basert på en valgt justeringspoengmetode ved å tilordne en innledende global justering og deretter tilpasse sekvensundersett. De omjusterte delsettene blir deretter selv justert for å produsere den neste iterasjonens flere sekvensjustering. Ulike måter å velge sekvensundergrupper og objektivfunksjon gjennomgås i.
Motivfunn
Motivfunn, også kjent som profilanalyse, konstruerer globale flere sekvensjusteringer som prøver å justere korte konserverte sekvensmotiver mellom sekvensene i spørresettet. Dette gjøres vanligvis ved først å konstruere en generell global flersekvensjustering, hvoretter de svært bevarte områdene isoleres og brukes til å konstruere et sett med profilmatriser. Profilmatrisen for hver konserverte region er arrangert som en scoringsmatrise, men frekvensene for hver aminosyre eller nukleotid i hver posisjon er avledet fra den konserverte regionens karakterfordeling i stedet for fra en mer generell empirisk fordeling. Profilmatrisene brukes deretter til å søke i andre sekvenser etter forekomster av motivet de karakteriserer. I tilfeller der det originale datasettet inneholdt et lite antall sekvenser, eller bare svært beslektede sekvenser, blir pseudokontoer lagt til for å normalisere tegnfordelingene som er representert i motivet.
Teknikker inspirert av informatikk
En rekke generelle optimaliseringsalgoritmer som vanligvis brukes i informatikk, har også blitt brukt på problemet med justering av flere sekvenser. Skjulte Markov -modeller har blitt brukt til å produsere sannsynlighetspoeng for en familie av mulige flere sekvensjusteringer for et gitt spørresett; selv om tidlige HMM-baserte metoder produserte underveldende ytelse, har senere applikasjoner funnet dem spesielt effektive til å oppdage eksternt beslektede sekvenser fordi de er mindre utsatt for støy fra konservative eller semikonservative substitusjoner. Genetiske algoritmer og simulert annealing har også blitt brukt for å optimalisere flere sekvensjusteringspoeng, bedømt av en poengsumfunksjon som sum-of-pair-metoden. Mer fullstendige detaljer og programvarepakker finnes i hovedartikkelen flersekvensjustering .
De Burrows-Wheeler forvandle har blitt brukt til rask kort lese justering i populære verktøy som Bowtie og BWA. Se FM-indeks .
Strukturell justering
Strukturelle justeringer, som vanligvis er spesifikke for protein og noen ganger RNA -sekvenser, bruker informasjon om den sekundære og tertiære strukturen til proteinet eller RNA -molekylet for å hjelpe til med å justere sekvensene. Disse metodene kan brukes for to eller flere sekvenser og produserer vanligvis lokale justeringer; Fordi de er avhengig av tilgjengeligheten av strukturell informasjon, kan de imidlertid bare brukes for sekvenser hvis tilsvarende strukturer er kjent (vanligvis gjennom røntgenkrystallografi eller NMR-spektroskopi ). Fordi både protein- og RNA -strukturen er mer evolusjonært konservert enn sekvens, kan strukturelle justeringer være mer pålitelige mellom sekvenser som er veldig fjernt beslektede og som har divergerte såpass mye at sekvenssammenligning ikke pålitelig kan oppdage deres likhet.
Strukturelle justeringer brukes som "gullstandarden" for å evaluere justeringer for homologibasert proteinstrukturforutsigelse fordi de eksplisitt justerer områder av proteinsekvensen som er strukturelt like i stedet for å stole utelukkende på sekvensinformasjon. Imidlertid kan tydelig strukturelle justeringer ikke brukes i strukturforutsigelser fordi minst én sekvens i spørresettet er målet som skal modelleres, for hvilken strukturen ikke er kjent. Det har blitt vist at, gitt den strukturelle justeringen mellom et mål og en mal -sekvens, kan det produseres svært nøyaktige modeller av målproteinsekvensen; en stor snublestein i homologibasert strukturforutsigelse er produksjon av strukturelt nøyaktige justeringer gitt bare sekvensinformasjon.
DALI
DALI-metoden, eller avstandsmatriselinjering , er en fragmentbasert metode for å konstruere strukturelle justeringer basert på kontaktlikhetsmønstre mellom påfølgende heksapeptider i spørresekvensene. Den kan generere parvis eller flere justeringer og identifisere en spørresekvens strukturelle naboer i Protein Data Bank (PDB). Det har blitt brukt til å konstruere FSSP strukturell justeringsdatabase (Fold-klassifisering basert på struktur-struktur-justering av proteiner, eller familier med strukturelt lignende proteiner). Du kan få tilgang til en DALI -webserver på DALI, og FSSP ligger på The Dali Database .
SSAP
SSAP (sequential structure alignment program) er en dynamisk programmeringsbasert metode for strukturell justering som bruker atom-til-atom-vektorer i strukturrom som sammenligningspunkter. Den har blitt forlenget siden den opprinnelige beskrivelsen til å omfatte flere så vel som parvise justeringer, og har blitt brukt i konstruksjonen av CATH (klasse, arkitektur, topologi, homologi) hierarkisk databaseklassifisering av proteinfoldinger. CATH -databasen er tilgjengelig ved CATH Protein Structure Classification .
Kombinatorisk forlengelse
Den kombinatoriske forlengelsesmetoden for strukturell justering genererer en parvis strukturell justering ved å bruke lokal geometri for å justere korte fragmenter av de to proteinene som analyseres, og deretter samler disse fragmentene til en større justering. Basert på tiltak som gjennomsnittlig kvadratisk avstand fra stiv kroppsrot , restavstander, lokal sekundærstruktur og omgivende miljøfunksjoner som hydrofobisitet av rester , genereres lokale justeringer kalt "justerte fragmentpar" og brukes til å bygge en likhetsmatrise som representerer alle mulige strukturelle justeringer innenfor forhåndsdefinerte grenseverdier. En vei fra en proteinstrukturtilstand til den andre spores deretter gjennom matrisen ved å forlenge den voksende justeringen ett fragment om gangen. Den optimale slike banen definerer den kombinatoriske forlengelsen. En nettbasert server som implementerer metoden og tilbyr en database med parvise justeringer av strukturer i Protein Data Bank, er lokalisert på Combinatorial Extension- nettstedet.
Fylogenetisk analyse
Filogenetikk og sekvensjustering er nært beslektede felt på grunn av den felles nødvendigheten av å evaluere sekvensrelatert. Feltet av phylogenetics gjør utstrakt bruk av sekvenssammenstillinger i konstruksjon og tolkning av fylogenetiske trær , som brukes til å klassifisere de evolusjonære forholdet mellom homologe gener representert i genomene av avvikende art. I hvilken grad sekvenser i et spørresett er forskjellige, er kvalitativt relatert til sekvensens evolusjonære avstand fra hverandre. Grovt sett antyder høy sekvensidentitet at de aktuelle sekvensene har en relativt ung siste felles stamfar , mens lav identitet tyder på at divergensen er eldre. Denne tilnærmingen, som gjenspeiler hypotesen om " molekylær klokke " om at en omtrent konstant evolusjonær endring kan brukes til å ekstrapolere den forløpte tiden siden to gener først divergerte (det vil si koalescens -tiden ), antar at effekten av mutasjon og seleksjon er konstant på tvers av sekvenslinjer. Derfor tar det ikke hensyn til mulig forskjell mellom organismer eller arter i hastigheten på DNA -reparasjon eller mulig funksjonell bevaring av spesifikke regioner i en sekvens. (Når det gjelder nukleotidsekvenser, reduserer molekylærklokkehypotesen i sin mest grunnleggende form også forskjellen i akseptrate mellom stille mutasjoner som ikke endrer betydningen av et gitt kodon og andre mutasjoner som resulterer i at en annen aminosyre blir inkorporert i proteinet). Mer statistisk nøyaktige metoder gjør at evolusjonshastigheten på hver gren av det fylogenetiske treet kan variere, og gir dermed bedre estimater av koalescens -tider for gener.
Progressive multiple alignment -teknikker produserer nødvendigvis et fylogenetisk tre fordi de inkorporerer sekvenser i den voksende justeringen etter slektskap. Andre teknikker som samler flere sekvensjusteringer og fylogenetiske trær, scorer og sorterer trær først og beregner en multi-sekvensjustering fra treet med høyest poengsum. Vanlige metoder for fylogenetisk trekonstruksjon er hovedsakelig heuristiske fordi problemet med å velge det optimale treet, som problemet med å velge den optimale flersekvensjusteringen, er NP-vanskelig .
Vurdering av betydning
Sekvensjusteringer er nyttige i bioinformatikk for å identifisere sekvenslikhet, produsere fylogenetiske trær og utvikle homologimodeller for proteinstrukturer. Den biologiske relevansen av sekvensjusteringer er imidlertid ikke alltid klar. Justeringer antas ofte å gjenspeile en grad av evolusjonær endring mellom sekvenser som stammer fra en felles forfader; Imidlertid er det formelt mulig at konvergent evolusjon kan oppstå for å gi tilsynelatende likhet mellom proteiner som er evolusjonært urelaterte, men utfører lignende funksjoner og har lignende strukturer.
I databasesøk som BLAST kan statistiske metoder bestemme sannsynligheten for at en bestemt justering mellom sekvenser eller sekvensområder oppstår ved en tilfeldighet gitt størrelsen og sammensetningen av databasen som søkes. Disse verdiene kan variere betydelig avhengig av søkeområdet. Spesielt øker sannsynligheten for å finne en gitt justering ved en tilfeldighet hvis databasen bare består av sekvenser fra samme organisme som spørresekvensen. Gjentatte sekvenser i databasen eller spørringen kan også forvride både søkeresultatene og vurderingen av statistisk signifikans; BLAST filtrerer automatisk slike repeterende sekvenser i spørringen for å unngå tilsynelatende treff som er statistiske artefakter.
Metoder for statistisk signifikansestimering for gapede sekvensjusteringer er tilgjengelige i litteraturen.
Vurdering av troverdighet
Statistisk signifikans indikerer sannsynligheten for at en justering av en gitt kvalitet kan oppstå ved en tilfeldighet, men indikerer ikke hvor mye bedre en gitt justering er enn alternative justeringer av de samme sekvensene. Tiltak for tilpasningstroverdighet indikerer i hvilken grad de best scorende justeringene for et gitt par sekvenser er vesentlig like. Metoder for justering av troverdighetsestimering for gapede sekvensjusteringer er tilgjengelige i litteraturen.
Poengfunksjoner
Valget av en poengsumfunksjon som gjenspeiler biologiske eller statistiske observasjoner om kjente sekvenser er viktig for å produsere gode justeringer. Proteinsekvenser justeres ofte ved hjelp av substitusjonsmatriser som gjenspeiler sannsynligheten for gitt karakter-til-tegn-substitusjoner. En serie matriser kalt PAM -matriser (Point Accepted Mutation matrices, opprinnelig definert av Margaret Dayhoff og noen ganger referert til som "Dayhoff -matriser") koder eksplisitt for evolusjonære tilnærminger til frekvenser og sannsynligheter for bestemte aminosyremutasjoner. En annen vanlig serie med scoringsmatriser, kjent som BLOSUM (Blocks Substitution Matrix), koder for empirisk avledede substitusjonssannsynligheter. Varianter av begge typer matriser brukes til å oppdage sekvenser med forskjellige nivåer av divergens, slik at brukere av BLAST eller FASTA kan begrense søk til nærmere beslektede treff eller utvide for å oppdage flere divergerende sekvenser. Gap -straffer utgjør innføringen av et gap - på evolusjonsmodellen, en innsetting eller sletting -mutasjon - i både nukleotid- og proteinsekvenser, og derfor bør straffeverdiene være proporsjonale med den forventede frekvensen av slike mutasjoner. Kvaliteten på justeringene som produseres, avhenger derfor av kvaliteten på poengsummen.
Det kan være veldig nyttig og lærerikt å prøve den samme justeringen flere ganger med forskjellige valg for å score matrise- og/eller gapstraffverdier og sammenligne resultatene. Regioner der løsningen er svak eller ikke-unik kan ofte identifiseres ved å observere hvilke områder av justeringen som er robuste for variasjoner i justeringsparametere.
Andre biologiske bruksområder
Sekvensert RNA, for eksempel uttrykte sekvensmerker og mRNA i full lengde, kan justeres til et sekvensert genom for å finne hvor det er gener og få informasjon om alternativ spleising og RNA-redigering . Sekvensjustering er også en del av genommontering , der sekvenser justeres for å finne overlapping slik at det kan dannes konti (lange sekvensstrekninger). En annen bruk er SNP -analyse, der sekvenser fra forskjellige individer er justert for å finne enkelt basepar som ofte er forskjellige i en populasjon.
Ikke-biologiske bruksområder
Metodene som brukes for biologisk sekvensjustering har også funnet applikasjoner på andre felt, særlig innen naturlig språkbehandling og i samfunnsvitenskap, der Needleman-Wunsch-algoritmen vanligvis blir referert til som Optimal matching . Teknikker som genererer settet med elementer som ord vil bli valgt i generasjonsalgoritmer for naturlig språk, har lånt flere sekvensjusteringsteknikker fra bioinformatikk for å produsere språklige versjoner av datagenererte matematiske bevis. Innen historisk og komparativ lingvistikk har sekvensjustering blitt brukt for delvis å automatisere den komparative metoden som lingvister tradisjonelt rekonstruerer språk. Forretnings- og markedsføringsforskning har også brukt flere sekvensjusteringsteknikker for å analysere serier av kjøp over tid.
Programvare
En mer komplett liste over tilgjengelig programvare kategorisert etter algoritme og justeringstype er tilgjengelig i programvare for sekvensjustering , men vanlige programvareverktøy som brukes til generelle sekvensjusteringsoppgaver inkluderer ClustalW2 og T-kaffe for justering, og BLAST og FASTA3x for databasesøk. Kommersielle verktøy som DNASTAR Lasergene , Geneious og PatternHunter er også tilgjengelige. Verktøy som er merket for å utføre sekvensjustering, er oppført i bio.tools -registret .
Justeringsalgoritmer og programvare kan sammenlignes direkte med hverandre ved hjelp av et standardisert sett av referansereferanseflersekvenssammenstillinger som er kjent som BAliBASE. Datasettet består av strukturelle justeringer, som kan betraktes som en standard som rent sekvensbaserte metoder sammenlignes mot. Den relative ytelsen til mange vanlige justeringsmetoder for ofte oppståtte justeringsproblemer har blitt tabellert og utvalgte resultater publisert online på BAliBASE. En omfattende liste over BAliBASE -score for mange (for tiden 12) forskjellige justeringsverktøy kan beregnes innenfor protein -arbeidsbenken STRAP.
Se også
- Sekvenshomologi
- Sekvensgruvedrift
- BLAST
- Stringsøkingsalgoritme
- Justeringsfri sekvensanalyse
- UGENE
- Needleman - Wunsch algoritme
- Smith-Waterman alogritme
Referanser
Eksterne linker
-
 Medier relatert til sekvensjustering på Wikimedia Commons
Medier relatert til sekvensjustering på Wikimedia Commons